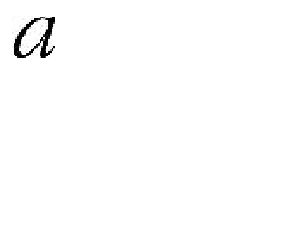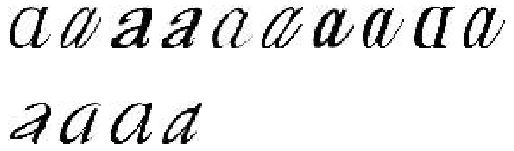Python计算机视觉编程第六章——图像聚类(K-means聚类,DBSCAN聚类,层次聚类,谱聚类,PCA主成分分析)
Python计算机视觉编程
- 图像聚类
- (一)K-means 聚类
- 1.1 SciPy 聚类包
- 1.2 图像聚类
- 1.1 在主成分上可视化图像
- 1.1 像素聚类
- (二)层次聚类
- (三)谱聚类
图像聚类
聚类概念
无监督学习:没有标签。(对于监督学习问题中,我们会被告知什么是正确答案,在无监督学习中,没有任何标签,或者都具有相同的标签,得到的数据如下图,图上有一系列点,但是它们没有标签,因此训练集可以写成 { x ( 1 ) , x ( 1 ) , x ( 1 ) , . . . , x ( m ) } \left \{ x^{(1)},x^{(1)},x^{(1)},...,x^{(m)} \right \} {x(1),x(1),x(1),...,x(m)},没有标签y,在无监督学习中,我们要将这系列无标签的数据输入到算法中,然后让算法找到一些隐含在数据中的结构,比如这个数据集中的点,可以分成两组分开的点集(簇),这种能够找到这些簇的算法称为聚类算法)

聚类:相似的东西分到一组。


可以看到这个图,在原始的数据集上是没有颜色的标记,没有告诉我们蓝色是哪个,绿色是哪个,红色是哪个,我们要把相似的东西归到一边,蓝色归到左下角,粉色归到右上角,也就是说根据数据分布的不同,把相似的往一边凑,凑完之后得到三个簇,也就是三个不同的类别。
难点:有监督问题,可以在预测值和真实值之间作比较,按某种方法评估出来一个指标,但是在无监督问题中,没有标签,评估就比较困难,还有就是调参,比如第一个参数得到一个数据结果,第二个参数得到一个数据结果,那第一个参数和第二个参数哪个好呢,很难比较,因为我们没有标签,不知道这两种情况分的怎么样,没有标准答案。
(一)K-means 聚类
定义:k-means算法中的k代表类簇个数,means代表类簇内数据对象的均值(这种均值是一种对类簇中心的描述),因此,k-means算法又称为k-均值算法。k-means算法是一种基于划分的聚类算法,以距离作为数据对象间相似性度量的标准,即数据对象间的距离越小,则它们的相似性越高,则它们越有可能在同一个类簇。
首先我们必须知道这几个概念:
1.要得到簇的个数,需要指定K值(k=2,输入的数据聚成2个堆;k=3,输入的数据聚成3个堆)
2.质心:数据的均值,即各个数据在各个维取平均值得到的值(例如现在有x轴,y轴,x轴上所有数据取均值,y轴所有数据取均值,这样我们就得到了一个质心。每个簇都可以得到一个质心,质心在后面迭代的时候要用到。)
3.距离的度量:聚类简单理解就是把相似的东西聚到一起,如何判断两个样本点是不是相似的呢,这个就要根据距离做判断,最常见的计算方式就是欧几里得距离(直接算两个点的欧式距离)和余弦相似度(先标准化),当使用欧式距离的时候,先要对数据进行标准化,
什么是标准化:比如现在有两个维度,x轴,y轴,x轴数据0.01,0.02,0.04,y轴数据100,200,300,当计算相似度的时候,x轴的差异无论怎么算都比较小,y轴的差异无论怎么算都比较大,那样我们潜意识里就认为相似度主要由y轴决定,实际算出来也是这样,所以说,在使用距离的度量的时候,基本情况下,都要对所有数据进行标准化,比如让x轴取值范围在0到1之间,y轴取值范围在0到1之间,让数据x和y基本在一个比较小的范围内浮动,比如说都是0到1,或者-1到1。
先把数据做标准化,然后再用距离的度量看一下什么样的两个样本点是相似的,再把相似的分到一簇。
4.优化目标:
m i n ∑ i = 1 k ∑ x ∈ C i d i s t ( c i , x ) 2 min\sum_{i=1}^{k}\sum_{x\in C_{i}}^{}dist(c_{i},x)^{2} mini=1∑kx∈Ci∑dist(ci,x)2
最外层i从1到k,表示一共有多少簇,k=3的时候,表示对3个簇分别进行优化,里面一层是针对对每一个簇来说,每一个样本点到质心的距离算出来,优化目标就是每一个样本点到质点的距离求和越小。为什么越小越好,因为越小越相似。我们的目的就是把相似的东西分到一组,假如现在图像有一个红色的点,这个点到红色簇质心的距离非常大,到蓝色簇质心的距离非常小,非常大是我们不希望看到的情况,所以在迭代优化的时候,把这个红色的点化成蓝色就好了。总的来说,就是对于每一个点,要划分到合适的地方,如何看合不合适,就看它到中心的距离,比如它到红色中心的距离比较大,到蓝色中心的距离比较小,所以说它是蓝色的可能性更大,就会把它规划到蓝色簇。
工作流程:
图解:
讲解:
- 由于是无监督问题,不知道每一个点该属于哪一个簇,假设k=2( k=2,分两堆;k=3,分三堆,此处以k=2举例 ),首先会在(a)图中初始化两个点。
- 如(b)图所示,一个黄色的,一个粉色的,这两个点是随机初始化的。然后基于初始化的这两个点,算每一个其他的点是属于粉色还是黄色,如何判断,就是算这个点到黄色点的距离(如下图),假设为 d 1 d_1 d1,这个点到红色点的距离,假设为 d 2 d_2 d2, d 1 d_1 d1< d 2 d_2 d2,认为当前点属于黄色,因为距离越小越相似。以此类推。把每一个样本点都算一遍。一些离黄色近的化成黄色,一些离粉色近的化成粉色。得到了( c )图。
- 可以看到c图离预期效果还有一段距离。所以要进行更新,更新衡量的依据,也就是两个质心,因为这两个质心是随机选择的,肯定是不太准的。如何更新,就是把所有黄色的点拿出来,重新算质心,所有粉色的点同样算质心。得到了(d)图。
- 质心更新完之后,重新遍历样本中所有的点,比如原来黄色的点到粉色质心的距离 d 2 d_2 d2更小(如下图),现在就更新到粉色。以此类推。得到了(e)图。
- 再按照之前的套路,更新质心,得到(f)图。
- 再进行遍历,找每个点的归属,然后不断的更新下去,直到更新到某一步,所有的样本点不再发生变化。不再发生变化,更新基本结束,因为再更新还是原来状态,黄色的还是属于黄色,粉色的还是属于粉色。
优势:
- 简单(原理很简单,理解起来也很简单)
- 快速(直接考虑k值就可以了)
- 适合常规数据集(比如上图,两堆看起来就能分开)
劣势:
- k值难确定(当拿到一个数据,可以做可视化展示,但是由于标签不知道是什么,所以不知道究竟分成几个簇,普遍情况下需要设置多组,然后看效果)。








可以看到,好像分成几个簇都能划分出来,所以到底分成多少个簇合适,这个比较难确定。(小插曲:可能会有疑惑,为什么第八张图和第九张图与前面的分布有点不一样,是因为博主有点强迫症哈,本来想用第七张图与第六章图对比一下就可以了,结果发现七张图不好排版,但是这时候博主已经关掉了这个页面,所以第二次运行,又会重新设置初值,不同的初值得到的结果是不一样的,算是误打误撞发现新大陆吧) - 复杂度与样本呈现线性相关(复杂度与样本个数相关,所有的样本都要和质心计算,如果与样本过多,计算越多,复杂度就相当高了)
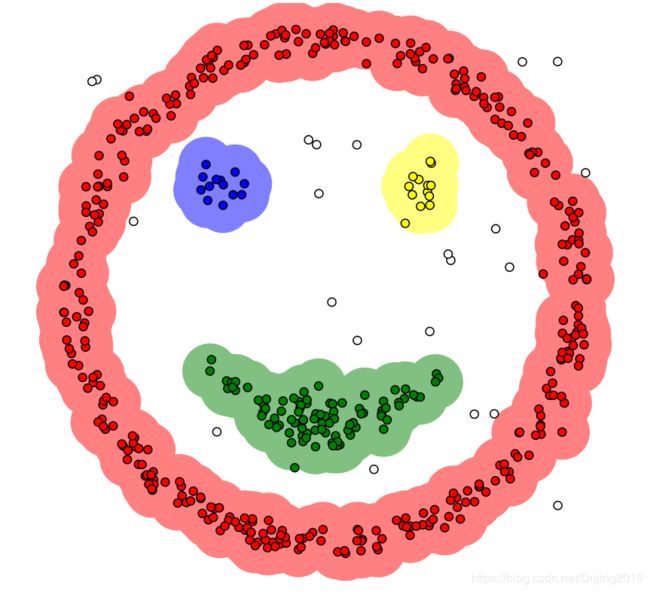
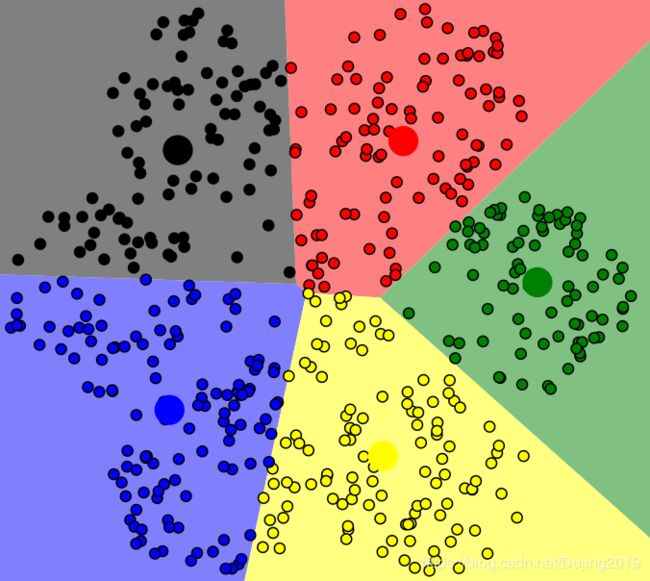
- 很难发现任意形状的簇(并不能保证一个数据是一个非常规整的簇,举例,如下图,可以认为里面是一个簇,外面是一个簇,但是用K-means算法没办法把里面和外面分开,而是会分为左边一个簇,右边一个簇)
下面来验证一下,这是一个笑脸,理想情况下应该分成两个眼睛,一个嘴巴,一个圆圈脸,四个簇,但是看下运行效果:




一直迭代,最后得到结果:

下面介绍K-means算法怎么实际运用在数据处理的。详细讲解见代码注释
# beer dataset
import pandas as pd
beer = pd.read_csv('data.txt', sep=' ') #导入数据,这是20个啤酒数据,包括四个属性
beer

X = beer[["calories","sodium","alcohol","cost"]] #聚类算法的输入是x,x是所有的特征,聚类算法不需要构建标签
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3).fit(X) #用三个堆做聚类
km2 = KMeans(n_clusters=2).fit(X) #用两个堆做聚类
km.labels_ #显示每个数据属于哪个类别,三个堆,所以有0,1,2
array([0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 0, 1, 0, 0, 0, 2, 0, 0, 2, 1])
km2.labels_ #两个堆,所以有0,1
array([1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 1, 0, 1, 1, 1, 0, 1, 1, 0, 0])
beer['cluster'] = km.labels_
beer['cluster2'] = km2.labels_
beer.sort_values('cluster')

from pandas.tools.plotting import scatter_matrix
%matplotlib inline
cluster_centers = km.cluster_centers_
cluster_centers_2 = km2.cluster_centers_
beer.groupby("cluster").mean() #分别算三个cluster的均值,观察不同类别上的差异性

beer.groupby("cluster2").mean() #分别算三个cluster的均值,观察不同类别上的差异性

centers = beer.groupby("cluster").mean().reset_index() #计算中心点
%matplotlib inline
import matplotlib.pyplot as plt
plt.rcParams['font.size'] = 14
import numpy as np
colors = np.array(['red', 'green', 'blue', 'yellow']) #指定绘图颜色
plt.scatter(beer["calories"], beer["alcohol"],c=colors[beer["cluster"]])
plt.scatter(centers.calories, centers.alcohol, linewidths=3, marker='+', s=300, c='black')#中心点
plt.xlabel("Calories")
plt.ylabel("Alcohol") #展示卡路里和酒精两个维度上

scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster"]], figsize=(10,10))
plt.suptitle("With 3 centroids initialized") ##展示四个维度

from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
X_scaled #标准化,使数据更规整

km = KMeans(n_clusters=3).fit(X_scaled)
beer["scaled_cluster"] = km.labels_
beer.sort_values("scaled_cluster") #标准化后得到的结果

beer.groupby("scaled_cluster").mean() #分别算归一化后三个cluster的均值,观察不同类别上的差异性

pd.scatter_matrix(X, c=colors[beer.scaled_cluster], alpha=1, figsize=(10,10), s=100) #显示四个维度

聚类评估:轮廓系数
s ( i ) = b ( i ) − a ( i ) m a x { a ( i ) , b ( i ) } s(i)=\frac{b(i)-a(i)}{max\left \{ a(i),b(i) \right \}} s(i)=max{a(i),b(i)}b(i)−a(i)
s ( i ) = { 1 − a ( i ) b ( i ) a ( i ) < b ( i ) 0 a ( i ) = b ( i ) b ( i ) a ( i ) − 1 a ( i ) > b ( i ) s(i)=\left\{\begin{matrix} 1-\frac{a(i)}{b(i)} & a(i)<b(i)\\ 0& a(i)=b(i)\\ \frac{b(i)}{a(i)}-1 & a(i)>b(i) \end{matrix}\right. s(i)=⎩⎪⎨⎪⎧1−b(i)a(i)0a(i)b(i)−1a(i)<b(i)a(i)=b(i)a(i)>b(i)
- 计算样本i到同簇其他样本的平均距离a(i)。a(i)越小,说明样本i越应该被聚类到该簇。将a(i)称为样本i的簇内不相似度。
- 计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:b(i) =min{bi1, bi2, …, bik},b(i)越大越好,越大说明当前样本越不可能被分到其他簇。
- si接近1,则说明样本i聚类合理。( b(i)-a(i)理想情况下等于b(i),max{a(i),b(i)}理想情况下等于b(i) )
- si接近-1,则说明样本i更应该分类到另外的簇
- 若si 近似为0,则说明样本i在两个簇的边界上。
from sklearn import metrics
score = metrics.silhouette_score(X,beer.cluster)#不做归一化得到的结果 si值
score_scaled = metrics.silhouette_score(X,beer.scaled_cluster)#归一化之后得到的结果 si值
print(score_scaled, score)#发现做完归一化结果反而低了,说明归一化有时候并不能使结果变好,比如某些特征确实很重要,那做完归一化就消除了差异
0.673177504646 0.179780680894
scores = []
for k in range(2,20): #遍历K值,看效果
labels = KMeans(n_clusters=k).fit(X).labels_
score = metrics.silhouette_score(X, labels)
scores.append(score)
scores #选择轮廓系数比较高的比较合适

plt.plot(list(range(2,20)), scores) #画图更直观展示
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Sihouette Score")

所以当k=2的时候合适。
下面介绍K-means算法怎么实际运用在图像处理的。
# -*- coding: utf-8 -*-
# 导包
from skimage import io
from sklearn.cluster import KMeans
import numpy as np
# 读取图片
image = io.imread("D:\\Python\\chapter6\\empire.jpg")
# 显示图片
io.imshow(image)
# 获取图片压缩前的信息
print ('image的类型:', type(image)) # numpy.ndarray类型
print ('image的尺寸:', image.shape) # 显示尺寸
print ('image的宽度:', image.shape[0]) # 图片宽度
print ('image的高度:', image.shape[1]) # 图片高度
print ('image的通道数:', image.shape[2]) # 图片通道数
print ('image的总像素个数:', image.size) # 显示总像素个数
print ('image的最大像素值:', image.max()) # 最大像素值
print ('image的最小像素值:', image.min()) # 最小像素值
print ('image的像素平均值:', image.mean()) # 像素平均值
# rows*cols*channel = 482*500*3
rows = image.shape[0]
cols = image.shape[1]
channel = image.shape[2]
# 样本数*channel = 241000*3
# 每个样本在不同通道都有存在1个点,即此时每个样本对应于3个点
image = image.reshape(image.shape[0] * image.shape[1], channel)
# 样本数*channel = 241000*1
# 使用Kmeans算法将3通道变为1通道(将原来很多的颜色用少量的颜色来表示),即此时每个样本只存在1个点
# n_clusters:K值,把集合分成K个簇;n_init:指定CPU个数;max_iter:最大的迭代次数
kmeans = KMeans(n_clusters=128, n_init=10, max_iter=200)
kmeans.fit(image)
clusters = np.asarray(kmeans.cluster_centers_, dtype=np.uint8)
# labels_:每个点的标签
labels = np.asarray(kmeans.labels_, dtype=np.uint8)
# rows*cols*channel = 482*500*1
labels = labels.reshape(rows, cols)
np.save('D:\\Python\\chapter6\\codebook_test.npy', clusters)
# 保存压缩后的图片
io.imsave('D:\\Python\\chapter6\\compressed_test.jpg', labels)
# 读取压缩后的图片
newimage = io.imread("D:\\Python\\chapter6\\compressed_test.jpg")
# 获取图片压缩后的信息
print ('newimage的类型:', type(newimage)) # numpy.ndarray类型
print ('newimage的尺寸:', newimage.shape) # 显示尺寸
print ('newimage的宽度:', newimage.shape[0]) # 图片宽度
print ('newimage的高度:', newimage.shape[1]) # 图片高度
# print ('newimage的通道数:', newimage.shape[2]) #图片通道数
print ('newimage的总像素个数:', newimage.size) # 显示总像素个数
print ('newimage的最大像素值:', newimage.max()) # 最大像素值
print ('newimage的最小像素值:', newimage.min()) # 最小像素值
print ('newimage的像素平均值:', newimage.mean()) # 像素平均值
# 显示压缩后的图片
io.imshow(newimage)
控制台输出:
(‘image\xe7\x9a\x84\xe7\xb1\xbb\xe5\x9e\x8b\xef\xbc\x9a’,
)
(‘image\xe7\x9a\x84\xe5\xb0\xba\xe5\xaf\xb8\xef\xbc\x9a’, (800L, 569L, 3L))
(‘image\xe7\x9a\x84\xe5\xae\xbd\xe5\xba\xa6\xef\xbc\x9a’, 800L)
(‘image\xe7\x9a\x84\xe9\xab\x98\xe5\xba\xa6\xef\xbc\x9a’, 569L)
(‘image\xe7\x9a\x84\xe9\x80\x9a\xe9\x81\x93\xe6\x95\xb0\xef\xbc\x9a’, 3L)
(‘image\xe7\x9a\x84\xe6\x80\xbb\xe5\x83\x8f\xe7\xb4\xa0\xe4\xb8\xaa\xe6\x95\xb0\xef\xbc\x9a’, 1365600)
(‘image\xe7\x9a\x84\xe6\x9c\x80\xe5\xa4\xa7\xe5\x83\x8f\xe7\xb4\xa0\xe5\x80\xbc\xef\xbc\x9a’, 255)
(‘image\xe7\x9a\x84\xe6\x9c\x80\xe5\xb0\x8f\xe5\x83\x8f\xe7\xb4\xa0\xe5\x80\xbc\xef\xbc\x9a’, 0)
(‘image\xe7\x9a\x84\xe5\x83\x8f\xe7\xb4\xa0\xe5\xb9\xb3\xe5\x9d\x87\xe5\x80\xbc\xef\xbc\x9a’, 132.4720262155829)
(‘newimage\xe7\x9a\x84\xe7\xb1\xbb\xe5\x9e\x8b\xef\xbc\x9a’,)
(‘newimage\xe7\x9a\x84\xe5\xb0\xba\xe5\xaf\xb8\xef\xbc\x9a’, (800L, 569L))
(‘newimage\xe7\x9a\x84\xe5\xae\xbd\xe5\xba\xa6\xef\xbc\x9a’, 800L)
(‘newimage\xe7\x9a\x84\xe9\xab\x98\xe5\xba\xa6\xef\xbc\x9a’, 569L)
(‘newimage\xe7\x9a\x84\xe6\x80\xbb\xe5\x83\x8f\xe7\xb4\xa0\xe4\xb8\xaa\xe6\x95\xb0\xef\xbc\x9a’, 455200)
(‘newimage\xe7\x9a\x84\xe6\x9c\x80\xe5\xa4\xa7\xe5\x83\x8f\xe7\xb4\xa0\xe5\x80\xbc\xef\xbc\x9a’, 167)
(‘newimage\xe7\x9a\x84\xe6\x9c\x80\xe5\xb0\x8f\xe5\x83\x8f\xe7\xb4\xa0\xe5\x80\xbc\xef\xbc\x9a’, 0)
(‘newimage\xe7\x9a\x84\xe5\x83\x8f\xe7\xb4\xa0\xe5\xb9\xb3\xe5\x9d\x87\xe5\x80\xbc\xef\xbc\x9a’, 60.489773725834795)
观察到Kmeans算法将3通道变为1通道,原来每个像素点取值是0到256,现在是0到167,从文件夹中查看图像属性发现压缩后图片的大小从215kb变成了150kb,Kmeans算法会把类似的颜色分别放在K个簇中——也就是说,每个簇的颜色都变成了一种。因此,我们只需要保留每个像素的标签(表明该像素在哪个簇中),以及每个簇的颜色编码即可完成图像的压缩。图像压缩效果比较好。
扩展:
DBSCAN聚类
基本概念:是一种基于密度的空间聚类算法。
核心对象:若某个点的密度达到算法设定的阈值则其为核心点。(即画一个圈,圈中点的个数多于阈值 ,也就是r 邻域内点的数量不小于 minPts(阈值))
ϵ-邻域的距离阈值:设定的半径r(DBSCAN算法需要指定两个参数,一个是半径,一个是阈值,不需要设置k值,究竟聚成多少堆,由算法决定)
直接密度可达:若某点p在点q的 r 邻域内,且q是核心点则p-q直接密度可达。

密度可达:若有一个点的序列q0、q1、…qk,对任意qi-qi-1是直接密度可达的 ,则称从q0到qk密度可达(如图,q0与q1直接密度可达,q1与q2直接密度可达,q0与q2密度可达),这实际上是直接密度可达的“传播”。

密度相连:若从某核心点p出发,点q和点k都是密度可达的 ,则称点q和点k是密度相连的。
边界点: 属于某一个类的非核心点,不能发展下线了。(比如:q0作为核心点发展了q1,q1作为核心点发展了q2,但是q2里面没有点了,则q2是边界点。)

噪声点:不属于任何一个类簇的点,从任何一个核心点出发都是密度不可达的。(例如:从任何一个核心点出发都不能把q3圈进来,)

工作流程:
讲解见注释
标记所有对象为unvisited
Do
随机选择一个 unvited 对象 p;
标记 p 为visited;
if p 的 ϵ-(半径范围内) 领域内至少有 MinPts 个对象 #如果p是核心对象
创建一个新簇 C,并把p添加到C;
令 N 为 p 的 ϵ- 领域中的对象集合
for N 中每个点 p #假设p里面有A,B,C,D四个点,则遍历这四个点,以A举例说明
if p 是 unvisited #如果p中的A没有被标记
标记 p 为visited #把P中的A标记为被访问过
if p 的ϵ-领域至少有 MinPts 个对象,把这些对象添加到N; #一开始p发展了A,B,C,D,现在A,B,C,D继续发展下线,都添加到N,直到所有发展的下线不是核心对象了
如果 p 还不是任何簇的成员,把 p 添加到 C; #把p,A,B,C,D以及它们的下线都放到C中,这样第一个簇就好了
End for;
输出 C;
Else 标记 p 为噪声;
Until 没有标记为unvisited 的对象。
参数选择:
- 半径ϵ:可以根据K距离(给定数据集P={p(i); i=0,1,…n},计算点P(i)到集合D的子集S中所有点之间的距离,距离按照从小到大的顺序排序,d(k)就被称为k-距离。)来设定:找突变点( P(i)到p(1)的距离d1,P(i)到p(2)的距离d2,P(i)到p(3)的距离d3,假设d1=0.1,d2=0.11,d3=0.12,而d4=0.3,d5=0.32,则d4就是突变点,根据这个突变点可以认为前面的这个距离(d=0.12)比较合适 )
(半径大,簇的个数可能就小了,半径小,簇的个数可能就多了,对结果有影响) - MinPts:点的个数,也就是密度。一般取的小一些,多次尝试。
优势:
-
不需要指定簇个数(基于密度,算法自己找)
-
擅长找到离群点(还有一些没有颜色的点就是离群点,不满足epsilon = 1.00,minPoints = 4,这是上图设置的两个参数。
) -
两个参数就够了
劣势:
- 高维数据处理有些困难(理论是这样,但是博主的实力现在还做不了这种实验,但是可以从另一个角度想,高维数据,点之间稀疏,密度就很难定义)
- 参数难以选择(参数对结果影响很大,如下图所示)
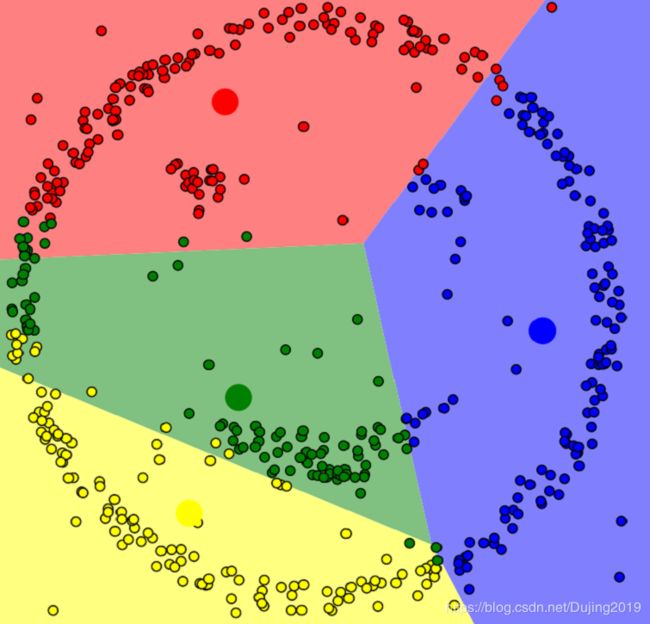
当epsilon = 1.00,minPoints = 4时

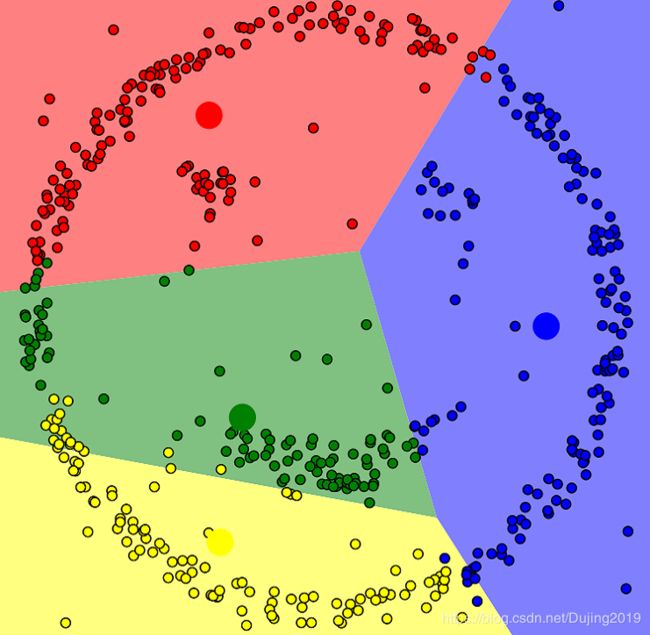
当epsilon = 0.92,minPoints = 2时

1.1 SciPy 聚类包
scipy.cluster是scipy下的一个做聚类的package, 共包含了两类聚类方法:
- 矢量量化(scipy.cluster.vq):支持vector quantization 和 k-means 聚类方法
- 层次聚类(scipy.cluster.hierarchy):支持hierarchical clustering 和 agglomerative clustering(凝聚聚类)
# coding=utf-8
from pylab import *
from scipy.cluster.vq import *
# 添加中文字体支持
from matplotlib.font_manager import FontProperties
font = FontProperties(fname=r"c:\windows\fonts\SimSun.ttc", size=14)
class1 = 1.5 * randn(100, 2)
class2 = randn(100, 2) + array([5, 5])
features = vstack((class1, class2))
centroids, variance = kmeans(features, 2) #计算方差
code, distance = vq(features, centroids)
figure()
ndx = where(code == 0)[0]
plot(features[ndx, 0], features[ndx, 1], '*')
ndx = where(code == 1)[0]
plot(features[ndx, 0], features[ndx, 1], 'r.')
plot(centroids[:, 0], centroids[:, 1], 'go')
title(u'2维数据点聚类', fontproperties=font)
axis('off')
show()

讲解:首先生成简单的二维数据,用 k=2 对这些数据进行聚类,由于 SciPy 中实现的 K-means 会计算若干次(默认为 20 次),并为我们选择方差最小的结果,所以这里返回的方差并不是我们真正需要的。用 SciPy 包中的矢量量化函数code,distance = vq(features,centroids)对每个数据点进行归类,为了将其可视化,可以画出这些数据点及最终的聚类中心,函数 where() 给出每个类的索引,绘制出的结果如上图所示。类中心标记为绿色大圆环,预测出的类分别标记为蓝色星号和红色点。
1.2 图像聚类
用 K-means 对这些字体图像进行聚类。
# -*- coding: utf-8 -*-
from PCV.tools import imtools
import pickle
from scipy import *
from pylab import *
from PIL import Image
from scipy.cluster.vq import *
from PCV.tools import pca
# Uses sparse pca codepath.
imlist = imtools.get_imlist('D:\\Python\\chapter6\\selectedfontimages\\a_selected_thumbs\\')
# 获取图像列表和他们的尺寸
im = array(Image.open(imlist[0])) # open one image to get the size
m, n = im.shape[:2] # get the size of the images
imnbr = len(imlist) # get the number of images
print "The number of images is %d" % imnbr
# Create matrix to store all flattened images
immatrix = array([array(Image.open(imname)).flatten() for imname in imlist], 'f')
# PCA降维
V, S, immean = pca.pca(immatrix)
# 保存均值和主成分
f = open('D:\\Python\\chapter6\\fontimages\\font_pca_modes.pkl', 'wb')
pickle.dump(immean, f)
pickle.dump(V, f)
f.close()
# 获取 selected-fontimages 文件下图像文件名,并保存在列表中
imlist = imtools.get_imlist('D:\\Python\\chapter6\\selectedfontimages\\a_selected_thumbs\\')
imnbr = len(imlist)
# 载入模型文件
with open('D:\\Python\\chapter6\\fontimages\\font_pca_modes.pkl', 'rb') as f:
immean = pickle.load(f)
V = pickle.load(f)
# 创建矩阵,存储所有拉成一组形式后的图像
immatrix = array([array(Image.open(im)).flatten() for im in imlist], 'f')
# 投影到前 40 个主成分上
immean = immean.flatten()
projected = array([dot(V[:40], immatrix[i] - immean) for i in range(imnbr)])
# 进行 k-means 聚类
projected = whiten(projected)
centroids, distortion = kmeans(projected, 4)
code, distance = vq(projected, centroids)
# 绘制聚类簇
for k in range(4):
ind = where(code == k)[0]
figure()
gray()
for i in range(minimum(len(ind), 40)):
subplot(4, 10, i + 1)
imshow(immatrix[ind[i]].reshape((25, 25)))
axis('off')
show()
1.1 在主成分上可视化图像
为了便于观察上面是如何利用主成分进行聚类的,我们可以在一对主成分方向的坐标上可视化这些图像。一种方法是将图像投影到两个主成分上,改变投影为:
projected = array([dot(V[[0,2]],immatrix[i]-immean) for i in range(imnbr)])
以得到相应的坐标(在这里 V[[0,2]] 分别是第一个和第三个主成分)。当然,也可以将其投影到所有成分上,之后挑选出需要的列。
# -*- coding: utf-8 -*-
from PCV.tools import imtools, pca
from PIL import Image, ImageDraw
from pylab import *
from PCV.clustering import hcluster
imlist = imtools.get_imlist('D:\\Python\\chapter6\\selectedfontimages\\a_selected_thumbs')
imnbr = len(imlist)
# Load images, run PCA.
immatrix = array([array(Image.open(im)).flatten() for im in imlist], 'f')
V, S, immean = pca.pca(immatrix)
# Project on 2 PCs.
projected = array([dot(V[[0, 1]], immatrix[i] - immean) for i in range(imnbr)])
# projected = array([dot(V[[1, 2]], immatrix[i] - immean) for i in range(imnbr)])
# 高和宽
h, w = 1200, 1200
# 创建一幅白色背景图
img = Image.new('RGB', (w, h), (255, 255, 255))
draw = ImageDraw.Draw(img)
# 绘制坐标轴
draw.line((0, h / 2, w, h / 2), fill=(255, 0, 0))
draw.line((w / 2, 0, w / 2, h), fill=(255, 0, 0))
# 缩放以适应坐标系
scale = abs(projected).max(0)
scaled = floor(array([(p / scale) * (w / 2 - 20, h / 2 - 20) + (w / 2, h / 2)
for p in projected])).astype(int)
# 粘贴每幅图像的缩略图到白色背景图片
for i in range(imnbr):
nodeim = Image.open(imlist[i])
nodeim.thumbnail((25, 25))
ns = nodeim.size
box = (scaled[i][0] - ns[0] // 2, scaled[i][1] - ns[1] // 2,
scaled[i][0] + ns[0] // 2 + 1, scaled[i][1] + ns[1] // 2 + 1)
img.paste(nodeim, box)
tree = hcluster.hcluster(projected)
hcluster.draw_dendrogram(tree, imlist, filename='fonts.png')
figure()
imshow(img)
axis('off')
img.save('D:\\Python\\chapter6\\pca_font.png')
show()
在上图成对主成分上投影的字体图像中。左图用的是第一个和第二个主成分,右图用的是第二个和第三个主成分。这里,我们用到了整数或 floor 向下取整除法运算符 //,通过移去小数点后面的部 分,可以返回各个缩略图在白色背景中对应的整数坐标位置。可以很清楚地看到,二维投影后相似的字体图像距离较近。
其实学到这,博主(小白)主成分是什么都不是很懂,所以在这个补一下主成分的知识。
主成分分析(PCA)
用途:降维中最常用的一种手段
目标:提取最有价值的信息(基于方差,找最大的方差方向,因为越大的方差方向,就会使数据降维后越分的开,数据分的开,就能更好的进行分类任务。)
问题:降维后的数据的意义不知道(只有机器知道,因为在数据变换中经过一系列指针变换,物理意义会变换没了,但是并不影响最后结果,但是我们也是想要最后结果,而不是阶段性目标)
向量的表示及基变换
向量的表示
内积: ( a 1 , a 2 , . . . , a n ) T . ( b 1 , b 2 , . . . , b n ) T = a 1 b 1 + a 2 b 2 + . . . + a n b n \left ( a_{1}, a_{2},...,a_{n}\right )^{T}.\left ( b_{1}, b_{2},...,b_{n}\right )^{T}=a_{1}b_{1}+a_{2}b_{2}+...+a_{n}b_{n} (a1,a2,...,an)T.(b1,b2,...,bn)T=a1b1+a2b2+...+anbn
解释: A ⋅ B = ∣ A ∣ ∣ B ∣ c o s ( a ) A\cdot B=|A||B|cos(a) A⋅B=∣A∣∣B∣cos(a)
设向量B的模为1,则A与B的内积值等于A向B所在直线投影的矢量长度。

B向量可以表示为(3,2),实际上表示线性组合: x ( 1 , 0 ) T + y ( 0 , 1 ) T x(1,0)^{T}+y(0,1)^{T} x(1,0)T+y(0,1)T
基变换
基:(1,0)和(0,1)叫做二维空间的一组基。
基是正交的(即内积为0,或直观说相互垂直)
要求:线性无关
变换:数据与一个基做内积运算,结果作为第一个新的坐标分量, 然后与第二个基做内积运算,结果作为第二个新坐标的分量
数据 (3,2) 映射到基中坐标:
( 1 / 2 1 / 2 − 1 / 2 1 / 2 ) ( 3 2 ) = ( 5 / 2 − 1 / 2 ) \begin{pmatrix} 1/\sqrt{2}& 1/\sqrt{2}\\ - 1/\sqrt{2}& 1/\sqrt{2} \end{pmatrix}\binom{3}{2}=\binom{5/\sqrt{2}}{-1/\sqrt{2}} (1/2−1/21/21/2)(23)=(−1/25/2)
( p 1 p 2 . . . p R ) ( a 1 a 2 . . . a M ) = ( p 1 a 1 p 1 a 2 . . . p 1 a M p 2 a 1 p 2 a 2 . . . p 2 a M . . . . . . . . . . . . . . . . . . p R a 1 p R a 2 . . . p R a M ) \begin{pmatrix} p_{1}\\ p_{2}\\ .\\ .\\ .\\ p_{R}\end{pmatrix}\begin{pmatrix} a_{1} & a_{2}& . & .& . & a_{M} \end{pmatrix}=\begin{pmatrix} p_{1}a_{1} & p_{1}a_{2} & . &. & . &p_{1}a_{M} \\ p_{2}a_{1} & p_{2}a_{2} & . &. & . &p_{2}a_{M}\\ .& . &. & .& . & .\\ .& . &. & .& . & .\\ .& . &. & .& . & .\\ p_{R}a_{1} & p_{R}a_{2} & . &. & . &p_{R}a_{M} \end{pmatrix} ⎝⎜⎜⎜⎜⎜⎜⎛p1p2...pR⎠⎟⎟⎟⎟⎟⎟⎞(a1a2...aM)=⎝⎜⎜⎜⎜⎜⎜⎛p1a1p2a1...pRa1p1a2p2a2...pRa2..................p1aMp2aM...pRaM⎠⎟⎟⎟⎟⎟⎟⎞
两个矩阵相乘的意义是将右边矩阵中的每一列列向量变换到左边矩阵中每一行行向量为基所表示的空间中去。
协方差矩阵
方向:如何选择这个方向(或者说基)才能尽量保留最多的原始信息呢,让数据维度降低,并且把主要成分都留下来。一种直观的看法是:希望投影后的投影值尽可能分散。
方差: V a r ( a ) = 1 m ∑ i = 1 m ( a i − u ) 2 Var(a)=\frac{1}{m}\sum_{i=1}^{m}(a_{i}-u)^{2} Var(a)=m1∑i=1m(ai−u)2 (表示一个特征分散的程度)
寻找一个一维基,使得所有数据变换为这个基上的坐标表示后,方差值最大
协方差(假设均值为0时):可以用两个字段的协方差表示其相关性
(表示a特征和b特征之间的关系,如果a和b变化趋势相同,则它们的协方差会很大,越接近1表示a和b的变化程度越大,协方差值在-1到1之间)
为什么假设均值为0:当我们拿到一份数据,通常情况下,我们要把数据以0为中心化,就是第一列减去第一列的均值,第二列减去第二列的均值,这样所有的数据都会以0为中心化。所以假设均值已经做了0中心化。
为什么引入协方差:如果单纯只选择方差最大的方向,后续方向应该会和方差最大的方向接近重合。(假如现在有一个10维的数据映射成2维的,假设现在找到一个轴,x轴,这个方向上方差最大,那方差次大的肯定是和x轴非常接近的一个轴,假设现在映射成4维,那y轴,z轴,w轴都会和x轴非常接近,接近重合,这时候就会出现问题,所有轴接近重合,那所有特征就接近相关,非常不利于建模,建模最好选择线性无关的)
解决方案:为了让两个字段尽可能表示更多的原始信息, 我们是不希望它们之间存在(线性)相关性的。可以用两个字段的协方差表示其相关性,当协方差为0时,表示两个字段完全独立.为了让协方差为0,选择第二个基时只能在与第一个基正交的方向上选择。因此最终选择的两个方向一定是正交的。
优化目标:
将一组N维向量降为K维(K大于0,小于N),目标是选择K个单位正交基,使原始数据变换到这组基上后,各字段两两间协方差为0,字段的方差则尽可能大。
协方差矩阵: X = ( a 1 a 2 . . . a m b 1 b 2 . . . b m ) X=\begin{pmatrix} a_{1} &a_{2} & . & . &. & a_{m}\\ b_{1}& b_{2} &. & . & . & b_{m} \end{pmatrix} X=(a1b1a2b2......ambm)
1 m X X T = ( 1 m ∑ i = 1 m a i 2 1 m ∑ i = 1 m a i b i 1 m ∑ i = 1 m a i b i 1 m ∑ i = 1 m b i 2 ) \frac{1}{m}XX^{T}=\begin{pmatrix} \frac{1}{m}\sum_{i=1}^{m}a_{i}^{2} &\frac{1}{m}\sum_{i=1}^{m}a_{i}b_{i} \\ \frac{1}{m}\sum_{i=1}^{m}a_{i}b_{i} & \frac{1}{m}\sum_{i=1}^{m}b_{i}^{2} \end{pmatrix} m1XXT=(m1∑i=1mai2m1∑i=1maibim1∑i=1maibim1∑i=1mbi2)
X是数据, X X T XX^T XXT相当于协方差矩阵,m是样本个数,矩阵对角线上的两个元素分别是两个字段的方差,而其它元素是a和b的协方差。
协方差矩阵对角化:即除对角线外的其它元素化为0,并且在对角线上 将元素按大小从上到下排列
协方差矩阵对角化: P C P T = Λ = ( λ 1 λ 2 . . . λ n ) PCP^{T}=\Lambda =\begin{pmatrix} \lambda _{1} & & & & & \\ & \lambda 2 & & & & \\ & & . & & & \\ & & &. & & \\ & & & & . & \\ & & & & & \lambda n \end{pmatrix} PCPT=Λ=⎝⎜⎜⎜⎜⎜⎜⎛λ1λ2...λn⎠⎟⎟⎟⎟⎟⎟⎞
实对称矩阵:一个n行n列的实对称矩阵一定可以找到n个单位正交特征向量
E = ( e 1 e 2 . . . e n ) E=\begin{pmatrix} e_{1} & e_{2} &... & e_{n} \end{pmatrix} E=(e1e2...en)
E T C E = Λ = ( λ 1 λ 2 . . . λ n ) E^{T}CE=\Lambda= \begin{pmatrix} \lambda _{1} & & & & & \\ & \lambda 2 & & & & \\ & & . & & & \\ & & &. & & \\ & & & & . & \\ & & & & & \lambda n \end{pmatrix} ETCE=Λ=⎝⎜⎜⎜⎜⎜⎜⎛λ1λ2...λn⎠⎟⎟⎟⎟⎟⎟⎞
根据特征值的从大到小(学到后面的时候,发现谱聚类是选择特征值小的,在这可以留意一下),将特征向量从上到下排列,则用前K行组成的矩阵乘以原始数据矩阵X,就得到了我们需要的降维后的数据矩阵Y。
举例:
数据: ( − 1 − 1 0 2 0 − 2 0 0 1 1 ) \begin{pmatrix} -1 & -1 & 0 & 2 &0 \\ -2& 0& 0 & 1 & 1 \end{pmatrix} (−1−2−10002101)
注:总共有五个数据,每个数据有两个特征,第一行为x1,第二行为x2,对x1,x2做以0为中心化,可以看到相加为0.
协方差矩阵: C = 1 5 ( − 1 − 1 0 2 0 − 2 0 0 1 1 ) ( − 1 − 2 − 1 0 0 0 2 1 0 1 ) = ( 6 5 4 5 4 5 6 5 ) C=\frac{1}{5}\begin{pmatrix} -1 & -1 & 0 & 2& 0\\ -2 & 0& 0 & 1 & 1 \end{pmatrix}\begin{pmatrix} -1 & -2\\ -1& 0\\ 0& 0\\ 2& 1\\ 0& 1 \end{pmatrix}=\begin{pmatrix} \frac{6}{5} & \frac{4}{5}\\ \frac{4}{5}& \frac{6}{5} \end{pmatrix} C=51(−1−2−10002101)⎝⎜⎜⎜⎜⎛−1−1020−20011⎠⎟⎟⎟⎟⎞=(56545456)
特征值: λ 1 = 2 , λ 2 = 2 / 5 \lambda _{1}=2,\lambda _{2}=2/5 λ1=2,λ2=2/5
特征向量: c 1 ( 1 1 ) , c 2 ( − 1 1 ) c_{1}\begin{pmatrix} 1\\ 1 \end{pmatrix},c_{2}\begin{pmatrix} -1\\ 1 \end{pmatrix} c1(11),c2(−11)
对角化: P C P T = ( 1 / 2 1 / 2 − 1 / 2 1 / 2 ) ( 6 / 5 4 / 5 4 / 5 6 / 5 ) ( 1 / 2 − 1 / 2 1 / 2 1 / 2 ) = ( 2 0 0 2 / 5 ) PCP^{T}=\begin{pmatrix} 1/\sqrt{2} &1/\sqrt{2} \\ -1/\sqrt{2}& 1/\sqrt{2} \end{pmatrix}\begin{pmatrix} 6/5 & 4/5\\ 4/5 & 6/5 \end{pmatrix}\begin{pmatrix} 1/\sqrt{2}& -1/\sqrt{2}\\ 1/\sqrt{2}& 1/\sqrt{2} \end{pmatrix}=\begin{pmatrix} 2 & 0\\ 0 & 2/5 \end{pmatrix} PCPT=(1/2−1/21/21/2)(6/54/54/56/5)(1/21/2−1/21/2)=(2002/5)
降维: Y = ( 1 / 2 1 / 2 ) ( − 1 − 1 0 2 0 − 2 0 0 1 1 ) = ( − 3 / 2 − 1 / 2 0 3 / 2 − 1 / 2 ) Y=\begin{pmatrix} 1/\sqrt{2} & 1/\sqrt{2} \end{pmatrix}\begin{pmatrix} -1 &-1 &0 &2 &0 \\ -2 & 0 & 0 & 1 & 1 \end{pmatrix}=\begin{pmatrix} -3/\sqrt{2} &-1/\sqrt{2} & 0 & 3/\sqrt{2} & -1/\sqrt{2} \end{pmatrix} Y=(1/21/2)(−1−2−10002101)=(−3/2−1/203/2−1/2)
pca在数据分析中的应用
假设原始数据有150个输入样本,有四个特征,那就是150x4的矩阵,进行特征压缩,变成2维,也就是150x2,所以要让原始数据乘以一个4x2的矩阵。所以接下来的目的就是怎么把4x2的矩阵构造出来。
from sklearn.preprocessing import StandardScaler
import numpy as np
import pandas as pd
df = pd.read_csv('iris.data')
X = df.ix[:,0:4].values
X_std = StandardScaler().fit_transform(X)
print (X_std) #这是一个四维数据
mean_vec = np.mean(X_std, axis=0)
cov_mat = (X_std - mean_vec).T.dot((X_std - mean_vec)) / (X_std.shape[0]-1) #对应下面公式
print('Covariance matrix \n%s' %cov_mat) #输出协方差矩阵
∑ = 1 n − 1 ( ( X − x ˉ ) T ( X − x ˉ ) ) \sum =\frac{1}{n-1}((X-\bar{x})^{T}(X-\bar{x})) ∑=n−11((X−xˉ)T(X−xˉ))
x ˉ = 1 n ∑ k = 1 n x i \bar{x}=\frac{1}{n}\sum_{k=1}^{n}x_{i} xˉ=n1∑k=1nxi
得到协方差矩阵:
Covariance matrix
[[ 1.00675676 -0.10448539 0.87716999 0.82249094]
[-0.10448539 1.00675676 -0.41802325 -0.35310295]
[ 0.87716999 -0.41802325 1.00675676 0.96881642]
[ 0.82249094 -0.35310295 0.96881642 1.00675676]]
cov_mat = np.cov(X_std.T)
eig_vals, eig_vecs = np.linalg.eig(cov_mat)
print('Eigenvectors \n%s' %eig_vecs) #特征向量
print('\nEigenvalues \n%s' %eig_vals) #特征值
特征值和特征向量一一对应的关系
Eigenvectors
[[ 0.52308496 -0.36956962 -0.72154279 0.26301409]
[-0.25956935 -0.92681168 0.2411952 -0.12437342]
[ 0.58184289 -0.01912775 0.13962963 -0.80099722]
[ 0.56609604 -0.06381646 0.63380158 0.52321917]]
Eigenvalues
[ 2.92442837 0.93215233 0.14946373 0.02098259]
特征值可以当成衡量特征向量重要程度的指标。因为降维成二维数据,所以需要提取两个特征值出来,就是最大的两个特征值。
tot = sum(eig_vals)
var_exp = [(i / tot)*100 for i in sorted(eig_vals, reverse=True)] #归一化操作,把特征值映射成一个百分数
print (var_exp)
cum_var_exp = np.cumsum(var_exp) #后一个值加前面一个值
cum_var_exp
将特征值归一化:
[72.620033326920336, 23.147406858644135, 3.7115155645845164, 0.52104424985101538] #归一化后的结果,变成百分数,例如第一个值占了72%重要
array([ 72.62003333, 95.76744019, 99.47895575, 100. ])
plt.figure(figsize=(6, 4))
plt.bar(range(4), var_exp, alpha=0.5, align='center',
label='individual explained variance')
plt.step(range(4), cum_var_exp, where='mid',
label='cumulative explained variance')
plt.ylabel('Explained variance ratio')
plt.xlabel('Principal components')
plt.legend(loc='best')
plt.tight_layout()
plt.show()
显示特征值以及累加结果:

柱形图是特征值,表示重要程度,线是特征值累加和,可以看出第一个和第二个特征向量比较重要,因为对应特征值比较大。
matrix_w = np.hstack((eig_pairs[0][1].reshape(4,1),
eig_pairs[1][1].reshape(4,1))) #先找第一大特征向量,再找第二大特征向量,组合到一块,成为4x2矩阵
print('Matrix W:\n', matrix_w) #变换矩阵
所以提取第一个和第二个特征向量,得到变换矩阵(也就是最开始要求的4x2矩阵):
Matrix W:
[[ 0.52308496 -0.36956962]
[-0.25956935 -0.92681168]
[ 0.58184289 -0.01912775]
[ 0.56609604 -0.06381646]]
Y = X_std.dot(matrix_w) #最终得到150x2矩阵
Y
plt.figure(figsize=(6, 4))
for lab, col in zip(('Iris-setosa', 'Iris-versicolor', 'Iris-virginica'),
('blue', 'red', 'green')):
plt.scatter(X[y==lab, 0],
X[y==lab, 1],
label=lab,
c=col)
plt.xlabel('sepal_len')
plt.ylabel('sepal_wid')
plt.legend(loc='best')
plt.tight_layout()
plt.show()
plt.figure(figsize=(6, 4))
for lab, col in zip(('Iris-setosa', 'Iris-versicolor', 'Iris-virginica'),
('blue', 'red', 'green')):
plt.scatter(Y[y==lab, 0],
Y[y==lab, 1],
label=lab,
c=col)
plt.xlabel('Principal Component 1')
plt.ylabel('Principal Component 2')
plt.legend(loc='lower center')
plt.tight_layout()
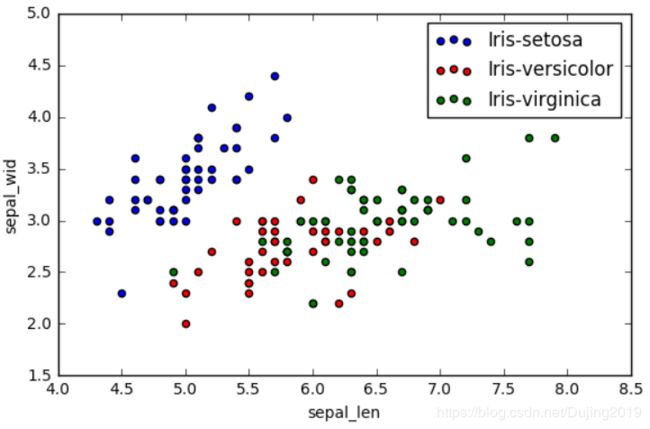
plt.show() #四维变换成两维
进行pca变换后,四维特征变换成两维,变换成两维后,红色点,蓝色点,绿色点分的更开了,比上图分辨能力更强了。但是x轴,y轴分别代表什么含义是不知道的,因为这是变换后的结果,很难来说它的物理意义,但是用它来进行分类是完全可以的,pca的效果还是不错的。

1.1 像素聚类
# -*- coding: utf-8 -*-
from scipy.cluster.vq import *
from scipy.misc import imresize
from pylab import *
from PIL import Image, ImageFont
# 添加中文字体支持
from matplotlib.font_manager import FontProperties
font = FontProperties(fname=r"c:\windows\fonts\SimSun.ttc", size=14)
def clusterpixels(infile, k, steps):
im = array(Image.open(infile))
dx = im.shape[0] / steps
dy = im.shape[1] / steps
# compute color features for each region
features = []
for x in range(steps):
for y in range(steps):
R = mean(im[x * dx:(x + 1) * dx, y * dy:(y + 1) * dy, 0])
G = mean(im[x * dx:(x + 1) * dx, y * dy:(y + 1) * dy, 1])
B = mean(im[x * dx:(x + 1) * dx, y * dy:(y + 1) * dy, 2])
features.append([R, G, B])
features = array(features, 'f') # make into array
# 聚类, k是聚类数目
centroids, variance = kmeans(features, k)
code, distance = vq(features, centroids)
# create image with cluster labels
codeim = code.reshape(steps, steps)
codeim = imresize(codeim, im.shape[:2], 'nearest')
return codeim
k = 3
infile_empire = 'D:\\Python\\chapter6\\empire.jpg'
im_empire = array(Image.open(infile_empire))
infile_boy_on_hill = 'D:\\Python\\chapter6\\boy_on_hill.jpg'
im_boy_on_hill = array(Image.open(infile_boy_on_hill))
steps = (50, 100) # image is divided in steps*steps region
print steps[0], steps[-1]
# 显示原图empire.jpg
figure()
subplot(231)
title(u'原图', fontproperties=font)
axis('off')
imshow(im_empire)
# 用50*50的块对empire.jpg的像素进行聚类
codeim = clusterpixels(infile_empire, k, steps[0])
subplot(232)
title(u'k=3,steps=50', fontproperties=font)
# ax1.set_title('Image')
axis('off')
imshow(codeim)
# 用100*100的块对empire.jpg的像素进行聚类
codeim = clusterpixels(infile_empire, k, steps[-1])
ax1 = subplot(233)
title(u'k=3,steps=100', fontproperties=font)
# ax1.set_title('Image')
axis('off')
imshow(codeim)
# 显示原图empire.jpg
subplot(234)
title(u'原图', fontproperties=font)
axis('off')
imshow(im_boy_on_hill)
# 用50*50的窗口对empire.jpg的像素进行聚类
codeim = clusterpixels(infile_boy_on_hill, k, steps[0])
subplot(235)
title(u'k=3,steps=50', fontproperties=font)
# ax1.set_title('Image')
axis('off')
imshow(codeim)
# 用100*100的窗口对empire.jpg的像素进行聚类
codeim = clusterpixels(infile_boy_on_hill, k, steps[-1])
subplot(236)
title(u'k=3,steps=100', fontproperties=font)
axis('off')
imshow(codeim)
show()
K-means 的输入是一个有 steps×steps 行的数组,数组的每一行有 3 列,各列分别为区域块 R、G、B 三个通道的像素平均值。为可视化最后的结果 , 用 SciPy 的 imresize() 函数在原图像坐标中显示这幅 steps×steps 的图像。参数 interp 指定插值方法;采用最近邻插值法,以便在类间进行变换时不需要引入新的像素值。K-means 标签的次序是任意的(在这里的标签指最终结果中图像的颜色)。正如看到的,尽管利用窗口对它进行了下采样,但结果仍然是有噪声的。如果图像某些区域没有空间一致性,则很难将它们分开,如图中小男孩和草坪的图。
(二)层次聚类
层次法(Hierarchicalmethods)先计算样本之间的距离。每次将距离最近的点合并到同一个类。然后,再计算类与类之间的距离,将距离最近的类合并为一个大类。不停的合并,直到合成了一个类。其中类与类的距离的计算方法有:最短距离法,最长距离法,中间距离法,类平均法等。比如最短距离法,将类与类的距离定义为类与类之间样本的最短距离。
层次聚类算法根据层次分解的顺序分为:自下底向上和自上向下,即凝聚的层次聚类算法和分裂的层次聚类算法(agglomerative和divisive),也可以理解为自下而上法(bottom-up)和自上而下法(top-down)。分裂层次聚类采用的就是"自顶而下"的思想,先将所有的样本都看作是同一个簇,然后通过迭代将簇划分为更小的簇,直到每个簇中只有一个样本为止。凝聚层次聚类采用的是"自底向上"的思想,先将每一个样本都看成是一个不同的簇,通过重复将最近的一对簇进行合并,直到最后所有的样本都属于同一个簇为止。
在凝聚层次聚类中,判定簇间距离的两个标准方法就是单连接(single linkage)和全连接(complete linkage)。单连接,是计算每一对簇中最相似两个样本的距离,并合并距离最近的两个样本所属簇。全连接,通过比较找到分布于两个簇中最不相似的样本(距离最远),从而来完成簇的合并。还可以通过平均连接(average linkage)。使用平均连接时,合并两个簇所有成员间平均距离最小的两个簇。
优势
- 可以通过绘制树状图(dendrogram),帮助我们使用可视化的方式来解释聚类结果。
- 不需要事先指定簇的数量。
举例:基于全连接的凝聚层次聚类
主要包括下面几个步骤:
- 获取所有样本的距离矩阵
- 将每个数据点作为一个单独的簇
- 基于最不相似(距离最远)样本的距离,合并两个最接近的簇
- 更新样本的距离矩阵
- 重复2到4,直到所有样本都属于同一个簇为止。
编写代码
1.获取样本,随机产生5个样本,每个样本包含3个特征(x、y、z)。
import pandas as pd
import numpy as np
if __name__ == "__main__":
np.random.seed(1)
#设置特征的名称
variables = ["x","y","z"]
#设置编号
labels = ["s1","s2","s3","s4","s5","s6"]
#产生一个(6,3)的数组
data = np.random.random_sample([6,3])*10
#通过pandas将数组转换成一个DataFrame
df = pd.DataFrame(data,columns=variables,index=labels)
#查看数据
print(df)

2.获取所有样本的距离矩阵,通过SciPy来计算距离矩阵,计算每个样本间两两的欧式距离,将矩阵矩阵用一个DataFrame进行保存,方便查看。
from scipy.spatial.distance import pdist,squareform
#获取距离矩阵
'''
pdist:计算两两样本间的欧式距离,返回的是一个一维数组
squareform:将数组转成一个对称矩阵
'''
dist_matrix = pd.DataFrame(squareform(pdist(df,metric="euclidean")),
columns=labels,index=labels)
print(dist_matrix)
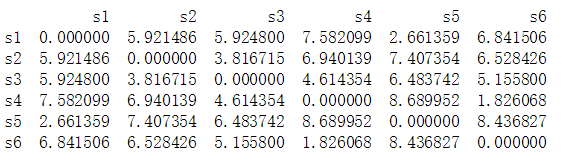
3.获取全连接矩阵的关联矩阵,通过scipy的linkage函数,获取一个以全连接作为距离判定标准的关联矩阵。
from scipy.cluster.hierarchy import linkage
#以全连接作为距离判断标准,获取一个关联矩阵
row_clusters = linkage(dist_matrix.values,method="complete",metric="euclidean")
#将关联矩阵转换成为一个DataFrame
clusters = pd.DataFrame(row_clusters,columns=["label 1","label 2","distance","sample size"],
index=["cluster %d"%(i+1) for i in range(row_clusters.shape[0])])
print(clusters)

第一列表示的是簇的编号,第二列和第三列表示的是簇中最不相似(距离最远)的编号,第四列表示的是样本的欧式距离,最后一列表示的是簇中样本的数量。
4.通过关联矩阵绘制树状图,使用scipy的dendrogram来绘制树状图。
from scipy.cluster.hierarchy import dendrogram
import matplotlib.pyplot as plt
row_dendr = dendrogram(row_clusters,labels=labels)
plt.tight_layout()
plt.ylabel("欧式距离")
plt.show()

通过上面的树状图,可以看到,首先是s1和s5合并,s4和s6合并,s2和s3合并,然后s2、s3、s4、s6合并,最后再和s1、s5合并。
层次聚类在图像处理中的应用
# -*- coding: utf-8 -*-
import os
from PCV.clustering import hcluster
from matplotlib.pyplot import *
from numpy import *
from PIL import Image
# 创建图像列表
path = 'D:\\Python\\chapter6\\sunsets\\flickr-sunsets-small\\'
imlist = [os.path.join(path, f) for f in os.listdir(path) if f.endswith('.jpg')]
# 提取特征向量,每个颜色通道量化成 8 个小区间
features = zeros([len(imlist), 512])
for i, f in enumerate(imlist):
im = array(Image.open(f))
# 多维直方图
h, edges = histogramdd(im.reshape(-1, 3), 8, normed=True, range=[(0, 255), (0, 255), (0, 255)])
features[i] = h.flatten()
tree = hcluster.hcluster(features)
# 设置一些(任意的)阈值以可视化聚类簇
clusters = tree.extract_clusters(0.23 * tree.distance)
# 绘制聚类簇中元素超过 3 个的那些图像
for c in clusters:
elements = c.get_cluster_elements()
nbr_elements = len(elements)
if nbr_elements > 3:
figure()
for p in range(minimum(nbr_elements, 20)):
subplot(4, 5, p + 1)
im = array(Image.open(imlist[elements[p]]))
imshow(im)
axis('off')
show()
hcluster.draw_dendrogram(tree, imlist, filename='sunset.pdf')
用100 幅日落图像进行层次聚类的示例聚类簇:


日落图像聚类后的树状图:

树状图的高和子部分由距离决定,随着坐标向下传递到下一级,会递归绘制出这些节点,上述代码用 20×20 像素绘制叶节点的缩略图,使用 get_height() 和 get_depth() 这两个辅助函数可以获得树的高和宽。最后展示了日落图像层次聚类后的树状图。可以看到,树中挨的相近的图像具有相似的颜色分布。
还可以对前面字体创建一个树状图:
tree = hcluster.hcluster(projected) hcluster.draw_dendrogram(tree,imlist,filename='fonts.jpg')

(三)谱聚类
谱聚类:是一种基于图论的聚类方法,通过对样本数据的拉普拉斯矩阵的特征向量进行聚类。它的本质是将聚类问题转化为图的最优划分问题。
图:由若干点及连接两点的线所构成的图像,通常用来描述某些事物之间的某种关系,用点代表事物,线表示对应两个事物间具有的关系。(有箭头是有向图,没有箭头是无向图,这个离散数学应该学过)
首先得到一些数据点,第一步,连点成线,抽象成简单的图的模型:
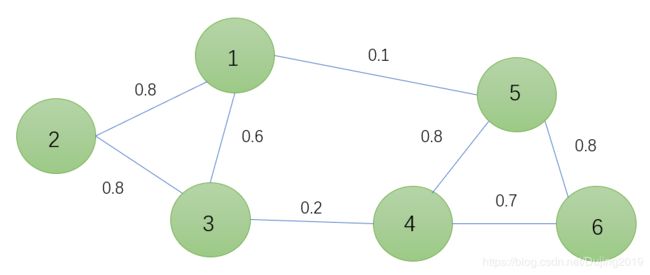
G ( V , E ) G(V,E) G(V,E)表示无向图(蓝色的线没有箭头), V = { v 1 , v 2 , . . . , v n } V=\left \{ v_{1},v_{2},...,v_{n} \right \} V={v1,v2,...,vn}表示点集(1,2,3,4,5,6这些点),E表示边集(蓝色的线)。
W i j W_{ij} Wij表示 V i V_i Vi与 V j V_j Vj之间的关系,称作权重(1到5之间的权重为0.1,5到1的权重也为0.1),对于无向图, W i j = W j i W_{ij}=W_{ji} Wij=Wji而且 W i i = 0 W_{ii}=0 Wii=0 (1到1本身权重为0) , W i j > 0 W_{ij}>0 Wij>0

划分时子图之间被截断的边的权重和(做聚类的时候一个很重要的指标):
C u t ( G 1 , G 2 ) = ∑ i ϵ G 1 , j ϵ G 2 W i j Cut(G_{1},G_{2})=\sum_{i\epsilon G_{1},j\epsilon G_{2}}^{}W_{ij} Cut(G1,G2)=iϵG1,jϵG2∑Wij
1,2,3聚类为 G 1 G_{1} G1,4,5,6聚类为 G 2 G_{2} G2,中间的权重0.1,0.2就是 W i j W_{ij} Wij
把权重列到表格中,可以看到权重矩阵是对角矩阵。该矩阵也叫邻接矩阵W(簇内部的各点以及到其他簇各点之间的邻接矩阵)。

把每一个节点到其他节点的度量相加,得到度矩阵D。

拉普拉斯矩阵 L = D − W L=D-W L=D−W
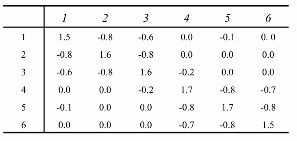
求出对应特征值,如果k=2,取前两个最小的特征值,那就是0.3和0.18,k=3,取前三个最小的特征值,那就是0.3和0.18,2.08。

找到这两个特征值对应的特征向量,重新形成一个矩阵U,横向看过去是每个样本点,纵向是两维的特征向量。

对这个特征向量形成的矩阵归一化,得到Y矩阵,再把每个点的坐标表示在图上。

再用kmeans可以形成两个聚类。

总的来说整个流程就是
- 数据准备,生成图的邻接矩阵
- 归一化拉普拉斯矩阵
- 生成最小的K个特征值和对应的特征向量
- 将特征向量kmeans聚类
优势:
- 处理稀疏数据(谱聚类只需要数据之间的邻接矩阵,因此对于处理稀疏数据的聚类很有效)
- 处理高维数据(使用了降维,因此在处理高维数据聚类时的复杂度比传统聚类算法好,在高维数据上表现明显)
劣势:
- 如果最终聚类的维度非常高,则由于降维的幅度不够,谱聚类的运行速度和最后的聚类效果均不好。
- 最终效果可能不同(聚类效果依赖于邻接矩阵,不同的邻接矩阵得到的最终聚类效果可能有很大不同)
下面用拉普拉斯矩阵的特征向量对字体图像进行谱聚类
# -*- coding: utf-8 -*-
from PCV.tools import imtools, pca
from PIL import Image, ImageDraw
from pylab import *
from scipy.cluster.vq import *
imlist = imtools.get_imlist('D:\\Python\\chapter6\\selectedfontimages\\a_selected_thumbs')
imnbr = len(imlist)
# Load images, run PCA.
immatrix = array([array(Image.open(im)).flatten() for im in imlist], 'f')
V, S, immean = pca.pca(immatrix)
# Project on 2 PCs.
projected = array([dot(V[[0, 1]], immatrix[i] - immean) for i in range(imnbr)]) # P131 Fig6-3左图
# projected = array([dot(V[[1, 2]], immatrix[i] - immean) for i in range(imnbr)]) # P131 Fig6-3右图
n = len(projected)
# 计算距离矩阵
S = array([[sqrt(sum((projected[i] - projected[j]) ** 2))
for i in range(n)] for j in range(n)], 'f')
# 创建拉普拉斯矩阵
rowsum = sum(S, axis=0)
D = diag(1 / sqrt(rowsum))
I = identity(n)
L = I - dot(D, dot(S, D))
# 计算矩阵 L 的特征向量
U, sigma, V = linalg.svd(L)
k = 5
# 从矩阵 L 的前k个特征向量(eigenvector)中创建特征向量(feature vector) # 叠加特征向量作为数组的列
features = array(V[:k]).T
# k-means 聚类
features = whiten(features)
centroids, distortion = kmeans(features, k)
code, distance = vq(features, centroids)
# 绘制聚类簇
for c in range(k):
ind = where(code == c)[0]
figure()
gray()
for i in range(minimum(len(ind), 39)):
im = Image.open(imlist[ind[i]])
subplot(4, 10, i + 1)
imshow(array(im))
axis('equal')
axis('off')
show()