NAR:UNITE真菌鉴定ITS数据库——处理未分类和并行分类(数据库文章阅读模板)
上方文章标题要求:包括杂志名缩写(一个词的杂志名不缩写,两个及上用实词首字母)和吸引人且有重要信息的语句,中间用中文冒号(:)分隔
注:文中灰色部分(引用格式)为写作说明文字,帮助编辑规范Markdown语言创造笔记和完成必要的内容,在正式稿件中可删除
目录[TOC],用于把握全文的层级结构,方便导航;一般分为三级标题;一级为文章标题,二级为摘要、背景、结果、方法和结论等,三级为主要结果、图表、方法中小节,内容较多的小节可设置四级标题。(此语法仅在有道云笔记/云协作/CSDN中支持较好,公众号发布时可删除)
文章目录
- 真菌分子鉴定联合数据库:处理暗分类和并行分类
- 划重点
- 摘要
- 背景
- 数据库
- 序列数据与质控
- UNITE物种注释
- 表1. 按98.5%相似度聚类各分类的地理信息(2018.8)
- 数据库结构和元数据标准
- 数据集
- 核心数据
- 辅助数据
- 用户导出数据集
- 网站
- 物种假设系统
- 图1. *Tomentella Atorarenicolor*在Unite SH数字对象标识符(DOI)页面的屏幕截图
- 鉴定服务
- 展望
- 参考文献
- 本文译者简介
- 猜你喜欢
- 写在后面
文章网页或PDF首页截图,包括杂志、标题、作者、单位等信息;具体以美观且有冲击力为主。推荐使用
FastCapture截图,图片命名为0.png
中英文标题,直译,中文为一级标题,英文加粗。注:以下全文所有段落之间保持一个空行作为段落分隔符。
真菌分子鉴定联合数据库:处理暗分类和并行分类
The UNITE database for molecular identification of fungi: handling dark taxa and parallel taxonomic classifications
译者和编辑姓名、单位,可使用笔名
翻译:刘永鑫 中科院遗传发育所
责编:刘永鑫 中科院遗传发育所
文章基本信息:杂志、影响因子、DOI、出版日期、(共同)第一作者、(共同)通讯作者、其它参与作者、一作和通讯单位或全部作者单位,注意上角标用文字实现
Nucleic Acids Research, [11.561]
https://doi.org/10.1093/nar/gky1022
Published online 29 October 2018
第一作者:Rolf Henrik Nilsson1
通讯作者:Rolf Henrik Nilsson1 [email protected]
合作作者: Karl-Henrik Larsson2, Andy F.S. Taylor3,
Johan Bengtsson-Palme 4,5,6, Thomas S. Jeppesen7, Dmitry Schigel7, Peter Kennedy8,
Kathryn Picard9, Frank Oliver Gl ¨ockner10, Leho Tedersoo11, Irja Saar11, Urmas K˜ oljalg11 and
Kessy Abarenkov12
主要单位(至少包括一作和通讯):
1 瑞典,哥德堡大学,生物与环境科学系,哥德堡全球生物多样性中心
划重点:译者总结文章要求,非摘要,根据背景提炼读者可能关注的信息,至少摘要、图表阅读后总结。提示:摘要中有重点信息,每个主图或表也有重要信息,软件可写用户体验。一般3-5句话,采用数字项目编号列表,200字左右。请先检索热心肠日报,有短科普可直接引用。
划重点
- UNITE数据库是目前真菌ITS整理最全面的数据库,基于上百万的全长ITS高质量序列,包括45万多个假定物种;
- 假定物种包括DOI号,方便在论文中引用提供唯一性;
- 新整合部分ITS2和PacBIO高通量数据,并改进末分类物种注释;
- 数据库为主流分析流程QIIME、Mothur和USEARCH都制作了相应格式的文件,方便使用。
摘要,原文翻译,尽量优化语言流畅度和可读性;注2, 3, 4级标题出现,需要在下方添加英文标题原文并加粗,方便读者查找阅读原文
摘要
ABSTRACT
UNITE(https://unite.ut.ee)是一个基于网络的真菌分子鉴定数据库和序列管理环境。它的目标是形成正式的真菌条形码——核核糖体内部转录间隔区(ITS)区域,并提供所有~1,000,000公共真菌ITS参考序列。这些被归类为~459,000个假定物种,并分配数字对象标识符(DOIs),以促进跨研究的精确参考引用。在过去15年中,内部和基于网络的第三方序列管理和注释对数据进行了超过275,000次改进。UNITE是一系列扩增子(宏条形码)分析流程的数据提供者,定期与所有主要真菌序列数据库和其他社区资源交换数据。最近的改进包括重新设计对不可分类假定物种的处理方案,整合了全球生物多样性信息设施的分类主体,以及支持不限制数量的并行计算分类系统。
软件、数据库类文章,摘要后截图主页或程序界面
UNITE数据库主页 https://unite.ut.ee
背景知识,专业词汇首次出来现,用括号注明英文原词,方便读者学习专业词汇;重点语句请加粗,方便读者重点阅读。注意链接结尾需要插入空格,保证链接正确结束
背景
INTRODUCTION
真菌界估计有2.2-3.8百万种的异养真核生物,DNA序列通常用于DNA条形码来探索真菌群落。大约600 bp的ITS是本领域的主要遗传标记,目前国际核苷酸合作序列数据库(International Nucleotide Sequence Databases Collaboration, INSDC)提供超过1,000,000的Sanger测序全长真菌ITS作为参考数据库。但这些序列用于为其它序列分类注释前,需要进行严格筛选和处理注释,因此2003年启动了真菌分子鉴定联合据库UNITE,致力于提供真菌的可重复鉴定,并通过收集和传播从其序列数据中得知的所有真菌的分类、生态和地理元数据,促进真菌学进展。
真菌的宏条形码与大量的OTU一直存在斗争。UNITE定期将序列按几个序列相似性阈值进行聚类,以获得假定物种(species hypotheses, SHs)—— 近似物种水平OTU。这些SHs分配有DOI,即使没有完整的名称也可以保持引用的统一性。大家可以下载多序列比对文件、使用blast比对SHs、使用最近基于概率分类的方法PROTAX、或其它序列搜索工具。此外,SHs提供了QIIME、MOTHUR、USEARCH和MICCA等主流软件兼容的数据格式文件 (https://unite.ut.ee/repository.php )。
UNITE支持基于网页的序列数据第三方注释,以反映最近的命名和分类变化,并纠正公共DNA序列中分类注释和其他元数据项的通常次优状态。通过组织针对性注释,例如植物病原真菌或构建真菌组,鼓励参与序列注释工作。所做的任何更改都是通过DOI的共同作者进行,由DataCite(https://www.datacite.org/ )监视的所有DOI(SHs)。UNITE面向社区的特性鼓励参与,但也包含了处理用户提供的注释审查过程。这一体系与一系列其他质量控制措施一起实施,以保持UNITE项目的高质量标准。
2003年,UNITE首次公开发行。从那时起,随着高通量测序(high-throughput sequencing, HTS)方法和认识到未被描述的“黑暗(dark,末被发现的)”类群渗透到真菌生命树中并可能主导地球的功能性生物多样性,真菌学发生了深远的变化。术语规则和分类原则的变化进一步推动了这一领域。在这篇文章中,我们详细介绍了我们为应对真菌和分子生态群落的技术和概念进步所带来的挑战而联合实施的最新应对方案。
数据库
DATABASES
序列数据与质控
标题保留英文原文,并加粗,方便读者快速定位原文段落阅读
Sequence data and quality control
公共真菌ITS序列来源于INDSC,并接受一系列质量控制措施,包括用ITSx和UCHIME处理,以尝试排除非ITS和嵌合体序列。不合格的条目将保留以备将来参考,但不用于鉴定的目的。发现包含ITS区域中完整ITS2子区域的序列以97-100%的相似性聚集在一起,以0.5%的步长生成SHs。从每个SH中最丰富的序列类型中随机选择一个代表性序列。目前正在努力将HTS研究产生的部分ITS序列纳入该系统,包括>10亿条真菌ITS序列的SRA数据库,再加上ITS1和ITS2序列的非重叠数据集难以关联阻碍了进展。我们目前正在努力将ITS2衍生的HTS研究添加到Unite中。与此同时,从PacBIO和Oxford Nanopore等技术中读取的长HTS正在逐渐变得可用。我们添加了第一组长PacBIO产生序列,包括完整的ITS区域和大约1000个碱基的LSU基因来联合和用于假定物种系统。
基于Web的第三方注释序列条目涵盖公共序列的大部分信息,包括分类名称、收集国家地区和收集条件。保留所有注释的历史记录,以便能够跟踪随时间的变化,例如在给定序列的名称中。允许表达分类注释的不同观点(competing views)。截至2018年秋季,UNITE用户共提供276,889条第三方注释(包括收集国家增加101,833条、收藏条件注释69,539条、分类重新注释23,410条)。
UNITE物种注释
UNITE taxonomy
文中图表出现时,标明对应图表编号并加粗,方便读者结果描述和图表对应阅读
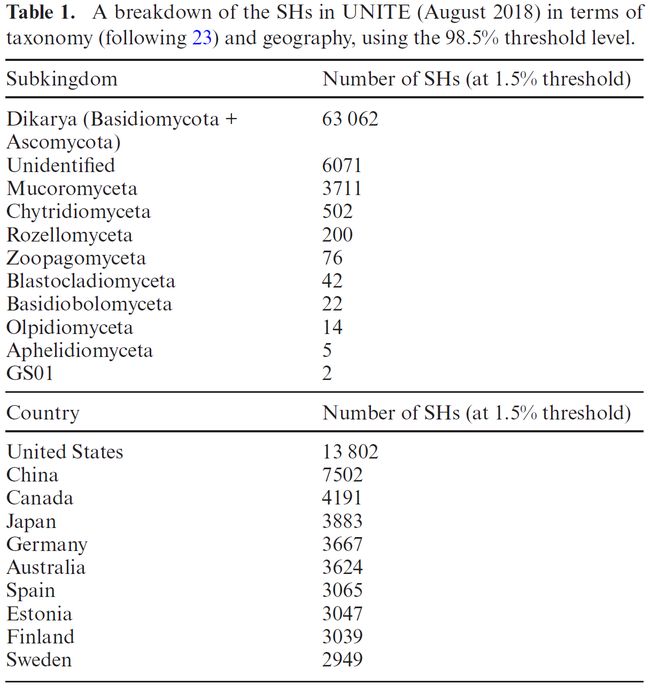
默认情况下,UNITE使用NCBI分类法分类作为分类主干,并辅以修改的Index Fungorum http://www.indexfungorum.org)和MycoBank为索引。表1提供了分类和地理方面的SHs细节(2018年8月)。UNITE的目标是向用户提供所有公共真菌的序列,从分类的角度来看,这些数据通常是复杂的,分类错误很普遍,大量缺乏有意义的分类注释的序列(例如“未培养的真菌”)。具有冲突分类信息序列的SHs由专家或经验丰富的用户标记为手动校正。此类受信任的第三方用户可以通过其Web浏览器重命名序列。来自类型材料的序列(例如GenBank RefSeq Loci)用于通知相似序列的分类注释。例如,一个未命名的序列在全球范围内至少有97%的相似性,它与一个完全注释的类型的序列对齐,这样可以在科水平安全地注释,通常也可以在属级别上注释。保留原始名称以及重新注释的历史记录,以供所有序列参考。
图表标题为三级标题,注释要全部翻译,位于图表下面
表1. 按98.5%相似度聚类各分类的地理信息(2018.8)
大多数SHs包括的序列来自多个国家,一些SH有由多国家贡献。图中仅列出了贡献最大的前10个国家。
虽然最近发表了一篇综述文章,但目前还没有公认的最新真菌分类系统。真菌分类处于不断变化的状态,不同的、部分不兼容的分类系统被用于不同的真菌资源,如Index Fungorum (http://www.index fungorum.org )、MycoBank (http://www.mycobank.org )和INDSC。为了满足存在竞争和分歧的分类系统导航的需要,UNITE 的新版本允许表示任意数量的可选分类系统。为此,我们引入了分类假设(Taxon Hypothesis, TH)的概念,允许基于序列的SHs在许多分类上同时通讯。与SHs一样,所有的THs都将收到基于DOI的唯一稳定标识符,并且单个SHs可以属于不同分类中的不同更高分类。不同研究的结果具有可比性,并且在使用DOIs进行通信时可重复使用。分类假设页面于2018年底上线。
数据库结构和元数据标准
Database structure and adherence to metadata standards
UNITE是在Plutof平台(https://plutof.ut.ee/ )上管理的数据集集合,使用PostgreSQL(https://www.postgresql.org/)和Postgis数据库引擎以及ember.js和drf框架。Plutof包含近200个表,并进行了建模,以允许准确、符合标准的DNA序列和序列元数据表示,如分类法、命名法、生态学和地理。对于序列数据和元数据,实现了MIXS标准。对于分类、生态和其他数据类型,使用了一系列标准(例如Darwincore、生态元数据语言和微生物学公共语言)。Plutof平台是数据站点联盟的一部分,该联盟允许UNITE发布SHs和DOI。Plutof平台与全球生物多样性信息设施(gbiodity information facility,GBIF;https://www.gbif.org/ )合作,UNITE的基于序列的SHs自2018年6月起成为GBIF分类骨干的一部分。这使得可以将宏条形码研究的结果直接上传到GBIF数据库(https://www.gbif.org/news/2lrgv5t3zugeu2wiymseuk )中,并将其作为生物学观察结果进行评分,即使基础分类群没有正式的科学名称。
数据集
Datasets
核心数据
UNITE core release
UNITE的序列数据以多种交互和静态方式提供给用户。我们的非冗余核心版本(https://unite.ut.ee/repository.php )包含来自每个非单体SH的代表性序列,目前包括458,797 SHs,以97–100%的相似性进行了不同的划分,以尽可能地反映物种水平,鉴于其在真菌生命之树内的变异性差异。此版本以fasta格式提供,例如本地BLAST搜索,以及为一系列扩增子分析流程(包括QIIME、MOTHUR和USEARCH)量身定制的格式。所有~1,000,000个序列的通用fasta版本也可用。经验丰富的用户将发现这些文件的其他可用变体,是否包括单体(singleton,只出现一次的序列)SHs、执行序列修剪的方式以及提供序列的分类隶属关系方面有所不同。所有的版本数据都包括分类的重新注释,这样它们与同一真菌ITS序列的INSDC版本有很大的不同。
辅助数据
UNITE auxiliary releases
UNITE提供了许多为满足科学界的特定需求而量身定制的发布数据集(https://unite.ut.ee/repository.php )。例如,“最需要的50种真菌”的数据包含了最大的SHs但目前没有任何有意义的分类注释,例如,仅门水平可以注释的序列。其目的是鼓励科学界澄清潜在分类群的分类学归属,并加快它们的正式特征和描述。由于用户的请求,我们还以JSON格式提供数据。我们很高兴考虑对其他格式和数据发布的请求。UNITE是GenBank的一个链接提供者,它将它们的全长真菌ITS序列链接到UNITE中相应的SH页面。自2018年以来,UNITE还是全球生物多样性信息设施(https://www.gbif.org/)的数据和链接提供商,这使得根据UNITE SH系统注释的真菌的序列可以被GBIF索引和查询。SHs成为第一个允许将分子检测和分子鉴定的生物多样性数据纳入GBIF全球数据池的系统,目前由自然历史收集和公民科学项目的形态学证据主导。除了科学使用外,全球政策制定和保护工作中还使用了由全球生物多样性基金(GBIF)介导的数据进行生物多样性评估。
用户导出数据集
Dataset exports by users
UNITE实现了一系列科学标准,这意味着用户可以定制几乎所有受这些标准控制的可想到的查询。例如,由于MIxS-BE和其他标准的原因,可以从建筑环境、在特定海拔或瑞典森林中收集的序列中下载所有序列。从蕨类植物、桉树属或水生环境中分离出的靶向序列同样简单,靶向病原真菌或人类相关真菌也是如此。可以使用多种格式进行下载,包括fasta、逗号分隔符格式,以及用户指定的导出类型。
网站
UNITE WEBSITE
UNITE网站(https://unite.ut.ee )提供对所有SHs、所有发布文件和所有元数据的直接访问。这些数据在CC BY-SA 4.0软件许可证下可用。下载数据或执行基本元数据和序列搜索不需要注册或登录。但是,要访问UNITE的高级功能,必须注册。注册是一个分层的过程,其中基本注册打开所有数据访问和下载功能。修改或存储数据需要额外的许可,并且一个复杂的系统已经就位,允许控制特定用户可能做什么,也可能不做什么。审查类型过程用于审查用户提供的数据和注释。注册用户可以申请成为特定分类群(如角叉菜科Cantharellaceae)的管理者,这使他们能够重新注释序列并为这些分类群提供新的元数据。要成为特定群体的版主,需要有文件证明你的分类专业知识。
物种假设系统
Species hypothesis system
所有SHs都有一个唯一的URL,其中显示基本的多序列比对和相关的元数据。每个SH都有一个独特的DOI,这使得它们可以在数据集和研究中进行交流,即使SH完全缺乏有意义的分类注释(例如“未培养的真菌”)。UNITE使用DOI版本控制系统,这样就可以随着时间的推移跟踪SH序列的包容性。因此,在某个特定的时间点,总是可以准确地确定哪些序列是某个特定SH的一部分。我们鼓励用户探索他们对我们系统感兴趣的真菌,这些真菌将序列和元数据集合在一起,并以INSDC无法实现的方式进行可视化(图1)。
图按顺序保存为1/2/N.png,图标题在图上为三级标题。图注在图下,每个子图注一段。对于多子图,可独立截图,尽量子图配图注,方便手机阅读和配合图注阅读。
图1. Tomentella Atorarenicolor在Unite SH数字对象标识符(DOI)页面的屏幕截图
https://plutof.ut.ee//datacite/10.15156%2Fbio%2Fsh009889.07fu
(A)从可用序列标识中自动选择(或手动选择,如果默认值被专家用户填写)。
(B)DOI的简短ID。
(C)选择参考序列的信息,以表示该SH。
(D)SH的真菌分类和单个序列的鉴定记录。Taxon名称后面的数字表示带有该名称的序列数。
(E)选择有关SH的统计数据。最小距离3.0%是姐妹SHs之间的强制遗传差异。
(F)单个序列的分布图。
(G)生态学信息(交互类群),如果与单个序列相关。
(H)DOI上数据站点的元数据。
(I)提取DNA的样本或样本的图像。只有有限数量的序列附有图像。
(J)SH的图形概述及详细信息。
(K)序列相似性阈值间的SH包容性。阈值(=最小距离)为1.5%,将这些序列分为两个SHs,以不同颜色显示。
(L)2.5%的阈值将所有序列合并为一个单独的SH。每个这样的SH都超链接到自己的唯一网页。
(M)SH的可滚动查看多序列比对。“RefSeq”表示手动选择该序列作为SHs的代表序列。
参考文献来源于类型样本或其他真实和特别值得信赖的材料。这个特殊的SH包含INSDC序列(棕色)和仅在UNITE(黄色)中发现的序列。在这个阶段,只有大约29,000个序列在UNITE中被发现,将在基础研究发表后提交给INSDC。这些序列包含在各种UNITE序列发布和下载文件中。
鉴定服务
Identification services
尽管UNITE主要是一个数据提供者,而不是一个宏条型码或序列分析软件流程,但我们确实为建立新产生的真菌ITS序列的分类隶属关系提供了基本手段。提供了一个多线程的BLAST服务,它是一个将序列集分配给SHs的功能。这些搜索享受UNITE社区提供的23000多个分类重新注释,这样UNITE中的查询将提供INSDC中不存在的信息。所有的重新注释都显示在原始数据中,尽管UNITE与其他序列数据库(包括INSDC)共享重新注释,但其中许多数据库在获得原始序列作者的书面同意之前,在不允许更改名称或序列的其他概念方面的政策下运行。
展望
OUTLOOK
单分子高通量测序技术的迅速发展,使产生跨越完整核糖体操纵子的高质量序列成为可能,包括SSU、ITS和LSU标记的全部。我们已经开始与SILVA数据库合作并交换结果,该数据库针对的是原核生物和真核生物SSU和LSU基因,而不是被夹在它们之间的真核生物ITS区域。相对保守的SSU和LSU基因提供了在门、类和目水平(通常更进一步)对新生成的序列数据进行强大的系统发育分配的优势,但代价是在物种、有时是属和科水平上的分辨率。改良的Unite-Silva合作将提供强大的系统发育位置和物种层面上的明确的可交流性,这是一种广受欢迎的组合。
UNITE将继续动态地向GBIF主干分类法提供SH分类法的版本,来自样本和宏条形码样本的分子出现数据也将通过 https://www.gbif.org 被发现。我们希望并期望将生物多样性的绘图扩展到分子领域,将推动更丰富和公正的生物多样性证据,从而对全球建模和决策产生积极影响。
UNITE致力于为真菌及其在真菌生命之树上的序列提供已解决的分类信息,并采用多种机制来确保这一点。然而,分类位置只能推断生物体生物学的一部分。最近,我们开始与FUNGuild数据库合作,提供关于UNITE中真菌种类和类群的功能性类别(guild )分配的信息。我们很快就可以展示这些真菌(例如菌根、动物寄生或腐生真菌)的分类名称,以及它们的功能组合。因此,使用者不仅可以获得一个分类学上的指纹,还可以获得手边真菌群落的功能/生态指纹。目前,Funguild在科、属和种名级别上与功能性分类联系在一起,这是一个潜在的问题,因为这些分类等级可以分别包括属、种或株,具有不同的生态生活方式。为了解决这个问题,UNITE将包括Funguild为每个任务提供的指定置信级别,以帮助用户进行解释。我们还预计能够将任务与单个DNA序列(真菌个体)联系起来,这样一个更紧密的联合真菌构建协作(所有数据都在两个方向上输入)将能够向UNITE和Funguild的用户提供有关真菌群落功能方面的高度详细信息。
UNITE数据库是一个开源的开放访问计划,由真菌学(Mycological)社区驱动。UNITE有一段适应真菌学界必须和需求的历史,我们的意思是保持这种方式。任何转发给UNITE工作人员的功能请求都将被考虑实施,所有这些实施都将免费提供给整个科学界。
参考文献
引文来自文章官网,包括所有作者的全名,杂志全名使用粗斜体,卷期年月和DOI链接(注:PubMed和Web of Science导出的引文名字和杂志都是缩写,用文章自己官网导出引文复制文本或导入Endnote,杂志Plant Cell格式即为全名)
Rolf Henrik Nilsson, Karl-Henrik Larsson, Andy F S Taylor, Johan Bengtsson-Palme, Thomas S Jeppesen, Dmitry Schigel, Peter Kennedy, Kathryn Picard, Frank Oliver Glöckner, Leho Tedersoo, Irja Saar, Urmas Kõljalg, Kessy Abarenkov, The UNITE database for molecular identification of fungi: handling dark taxa and parallel taxonomic classifications, Nucleic Acids Research, Volume 47, Issue D1, 08 January 2019, Pages D259–D264, https://doi.org/10.1093/nar/gky1022
本文译者简介
刘永鑫,博士。2008年毕业于东北农大微生物学专业。2014年中科院遗传发育所获生物信息学博士学位,2016年博士后出站留所工作,任宏基因组学实验室工程师,目前主要研究方向为宏基因组数据分析和植物微生物组。QIIME 2项目参与人,目前在***Nature Biotechnology、Plant Cell、Genomics Proteomics Bioinformatics、Science China Life Sciences***等杂志发表论文十余篇。2017年7月创办“宏基因组”公众号,目前分享宏基因组、扩增子原创文章600余篇,代表博文有《扩增子图表解读、分析流程和统计绘图三部曲(21篇)》、《QIIME2中文教程(18篇)》、《Nature综述:手把手教你分析菌群数据(1.8万字)》等,关注人数3.8万+,累计阅读500万+。
结尾的文章目录导航和二维码,每篇文章一致即可
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA