手把手教你R语言行NRI分析
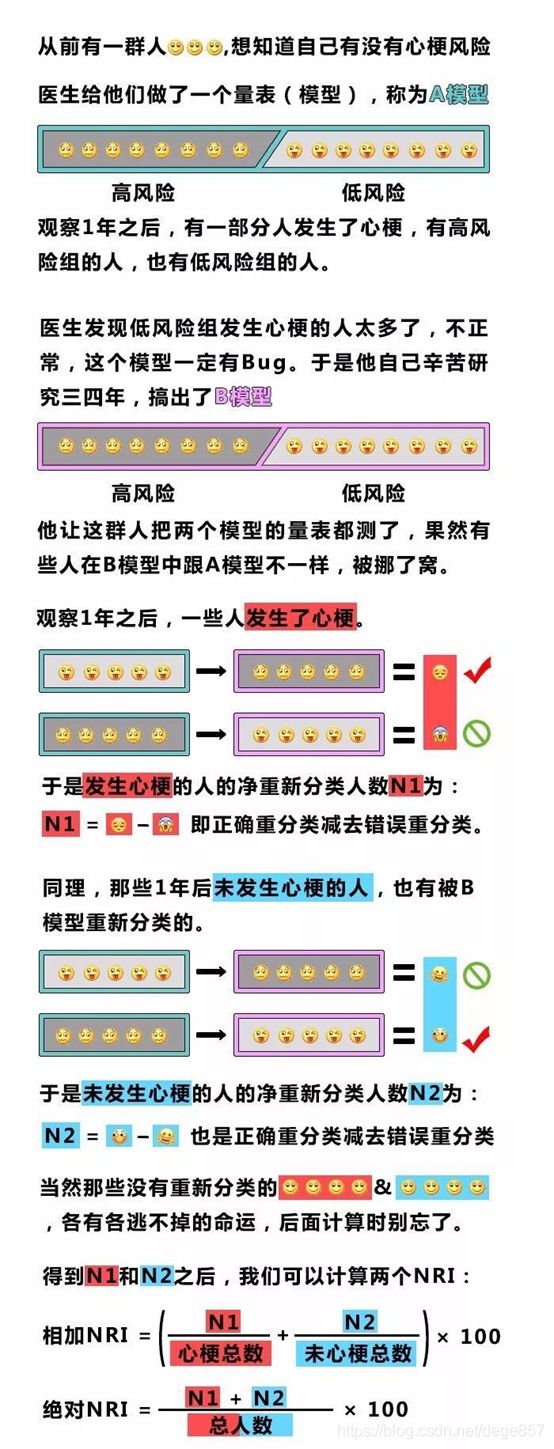
NRI(Net reclassification index)指净重新分类指数,用于我们在临床上比较新旧模型的效能,比如说:平时我们都用心电图评估心梗,现在有了新的指标肌钙蛋白,我们想了解肌酐蛋白联合心电图评估心梗和单用心电图哪个模型更好。我们知道一个模型建立后能产生一个切点,把一个人群分切阳性组和阴性组,新模型的建立后阳性组和阴性组肯定和原来模型不同,假阳性和假阴性也不同,NRI主要比较的是重新分布这类人群的真阳性和真阴性情况,原理我就不说了,说的也没别人说得好,看下面图片解释(图片来源于公众号:解螺旋)
NRI目前在SCI论文中应用越来越广,我们今天主要说说怎么使用R语言做出NRI,还是使用我们SPSS自带的乳腺癌的数据。
R语言做出NRI需要使用到nricens包和survival包,需要先下载好。我们先导入数据并查看变量。数据说明如下:
age表示年龄,pathsize表示病理肿瘤大小(厘米),lnpos表示腋窝淋巴结阳性,histgrad表示病理组织学等级,er表示雌激素受体状态,pr表示孕激素受体状态,status结局事件是否死亡,pathscat表示病理肿瘤大小类别(分组变量),ln_yesno表示是否有淋巴结肿大,time是生存时间,后面的agec是我们自己设定的,不用管它。
library(nricens)
library(survival)
library(foreign)
bc <- read.spss("E:/r/test/Breast cancer survival agec.sav",
use.value.labels=F, to.data.frame=T)
bc <- na.omit(bc)#删除缺失值
假如我们想知道48个月,也就是4年内的死亡情况,可以行COX回归分析
names(bc)
time<-bc$time#生成时间变量
status<-bc$status#生成结局变量
j1<-as.matrix(subset(bc,select = c(age,pathsize,pr,er)))#旧模型变量,要以矩阵形式,不能有缺失值,不然会报错
j2<-as.matrix(subset(bc,select = c(age,pathsize,pr,er,ln_yesno))) #新模型变量,要以矩阵形式,不能有缺失值,不然会报错
mod.std<-coxph(Surv(time,status)~ .,data.frame(time, status,j1),x=TRUE)#生成旧模型COX回归函数
mod.new<-coxph(Surv(time,status)~ .,data.frame(time, status,j2),x=TRUE) #生成新模型COX回归函数
p.std = get.risk.coxph(mod.std, t0=48)#生成预测函数
p.new = get.risk.coxph(mod.new, t0=48)#生成预测函数
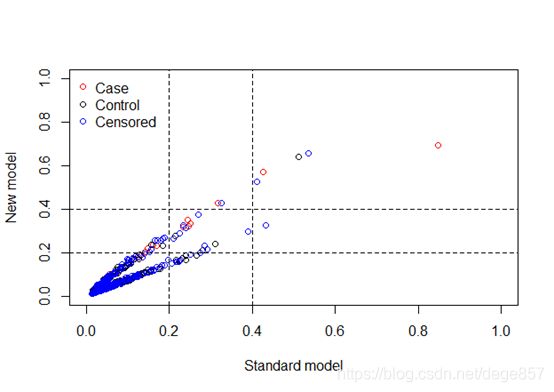
nricens(mdl.std = mod.std, mdl.new = mod.new, t0 = 48, cut = c(0.2, 0.4),
niter = 100,alpha = 0.05,updown = 'category')#cut分临床切点,分为高中低风险,niter为重抽样次数,category为分类的意思,alpha为置信区间


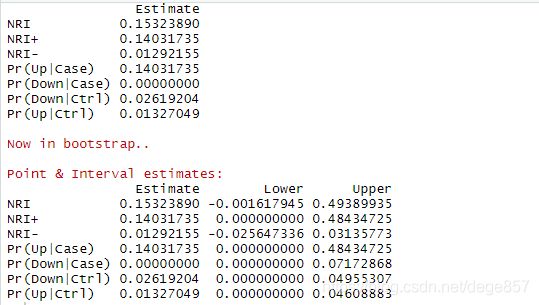
我们要关注NRI+这个值,它是正值表示,新模型优于旧模型,最后生成图表
也可以按分类变量及广义线性模型来处理,要先设置一下时间变量,和结局变量,其他基本一样的
bc= bc[ bc$time > 48| (bc$time < 48 & bc$status == 1), ]#重新定义一下数据
status1= ifelse(bc$time < 48 & bc$status == 1, 1, 0)#重新定义下结局变量
j3<-as.matrix(subset(bc,select = c(age,pathsize,pr,er)))
j4<-as.matrix(subset(bc,select = c(age,pathsize,pr,er,ln_yesno)))
mstd1= glm(status1 ~ ., binomial(logit), data.frame(status1, j3), x=TRUE)
mnew1= glm(status1 ~ ., binomial(logit), data.frame(status1, j4), x=TRUE)
nribin(mdl.std=mstd1, mdl.new = mnew1, cut = c(0.2, 0.4),
niter = 1000, updown = 'category')



