Python遗传和进化算法框架(一)Geatpy快速入门
Geatpy是一个高性能的Python遗传算法库以及开放式进化算法框架,由华南理工大学、华南农业大学、德州奥斯汀公立大学的学生联合团队开发。
- Website (including documentation): https://www.geatpy.com (repairing)
- Contact us: https://www.geatpy.com/supports
- Source: https://github.com/geatpy-dev/geatpy
- Bug reports: https://github.com/geatpy-dev/geatpy/issues
它提供了许多已实现的遗传和进化算法相关算子的库函数,如初始化种群、选择、交叉、变异、重插入、多种群迁移、多目标优化非支配排序等,并且提供开放式的进化算法框架来实现多样化的进化算法。其执行效率高于Matlab遗传算法工具箱和Matlab第三方遗传算法工具箱Gaot、gatbx、GEATbx,学习成本低。
Geatpy支持二进制/格雷码编码种群、实数值种群、整数值种群、排列编码种群。支持轮盘赌选择、随机抽样选择、锦标赛选择。提供单点交叉、两点交叉、洗牌交叉、部分匹配交叉(PMX)、线性重组、离散重组、中间重组等重组算子。提供简单离散变异、实数值变异、整数值变异、互换变异等变异算子。支持随机重插入、精英重插入。支持awGA、rwGA、i-awGA、nsga2、快速非支配排序等多目标优化的库函数、提供进化算法框架下的常用进化算法编程模板等。
关于遗传算法、进化算法的学习资料,在官网中https://www.geatpy.com (repairing)有详细讲解以及相关的学术论文链接。同时网上也有很多资料。
闲话少说……下面分享一下近几个星期以来的学习心得:
先说一下安装方法:
首先是要windows系统,Python要是3.5,3.6或3.7版本 ,并且安装了pip。只需在控制台执行
pip install geatpy
即可安装成功。或者到github上下载:https://github.com/geatpy-dev/geatpy 个人是推荐在github上下载。因为这样可以看到源码以及相关的demo程序,如果通过pip下载的话还需要找demo文件的位置,有些麻烦。
更新方法:
Geatpy在持续更新。可以通过以下命令使电脑上的版本与官方最新版保持一致:
pip install --upgrade
Geatpy提供2种方式实现遗传算法。先来讲一下第一种最基本的实现方式:编写编程脚本。
1. 编写脚本实现遗传算法:
用过谢菲尔德大学的Matlab遗传算法库Gatbx以及其升级版——GEATbx的朋友应该非常熟悉下面的Matlab脚本:
-
%% matlab_gatbx_test.m
-
-
%遗传算法求f(x)=x*sin(
10*pi*x)+
2.0,在[-
1,
2]上的最大值
-
figure(
1);
-
fplot(@(variable)variable.*sin(
10*pi*variable)+
2.0,[-
1,
2]); %画出函数曲线
-
tic %开始计时
-
%定义遗传算法参数
-
NIND=
40; %个体数目(Number of individuals)
-
MAXGEN=
25; %最大遗传代数(Maximum number of generations)
-
PRECI=
20; %变量的二进制位数(Precision of variables)
-
GGAP=
0.
9; %代沟(Generation gap)说明子代与父代的重复率为
0.
1
-
trace=zeros(MAXGEN,
2); %寻优结果的初始值
-
FieldD=[
20;-
1;
2;
1;
0;
1;
1]; %区域描述器(Build field descriptor),第
2、
3行为自变量的下界和上界
-
Chrom=crtbp(NIND, PRECI); %定义初始种群
-
gen=
0; %代计数器
-
variable=bs2rv(Chrom, FieldD); %计算初始种群的十进制转换
-
ObjV=shang(variable); %计算目标函数值
-
while gen
-
FitnV=ranking(-ObjV); %分配适应度值(Assign fitness values)ranking函数的功能就是目标值越小的分配值越大,
-
%本例求解最大值,应该要是他的适应度值更大,故必须使得ranking内的数越小,这样分配的适应度值才能大
-
SelCh=select(
'sus', Chrom, FitnV, GGAP); %选择,使用sus方式,也可以改用rws方式
-
SelCh=recombin(
'xovsp', SelCh,
0.
7); %重组,选用xovsp方式
-
SelCh=mut(SelCh); %变异
-
variable=bs2rv(SelCh, FieldD); %子代个体的十进制转换,把染色体变为十进制
-
ObjVSel=shang(variable); %计算子代的目标函数值
-
[Chrom ObjV]=reins(Chrom, SelCh,
1,
1, ObjV, ObjVSel); %重插入子代的新种群
-
variable=bs2rv(Chrom, FieldD); %子代个体的十进制转换,转为十进制
-
gen=gen+
1; %代计数器增加
-
%输出最优解及其序号,并在目标函数图像中标出,Y为最优解,I为种群的序号
-
[Y, I]=max(ObjV);hold on;
-
plot(variable(I), Y,
'ro');
-
trace(gen,
1)=max(ObjV); %遗传算法性能跟踪,把当代的最优值放入trace矩阵的第一行第目前代数列
-
trace(gen,
2)=sum(ObjV)/length(ObjV); %把当代种群目标函数的均值,放入trace矩阵的第二行第目前代数列
-
end
-
variable=bs2rv(Chrom, FieldD); %最优个体的十进制转换,转回十进制,以便输出
-
result = max(trace(
:,
1)) %输出搜索到的目标函数最大值
-
toc %结束计时
-
hold on;
-
grid on;
-
plot(variable,ObjV,
'b*');
-
figure(
2);
-
plot(trace(
:,
1)); %把trace矩阵的第一列画出来(记录的是每一代的最优值)
-
hold on;
-
plot(trace(
:,
2),
'-.');grid %把trace矩阵的第
2列画出来(记录的是每一代种群目标函数均值)
-
legend(
'解的变化',
'种群均值的变化')
-
-
-
function z=shang(x) % 目标函数的核心部分(即缺省了优化目标的纯函数)
-
z=x.*sin(
10*pi*x)+
2.0;
-
end
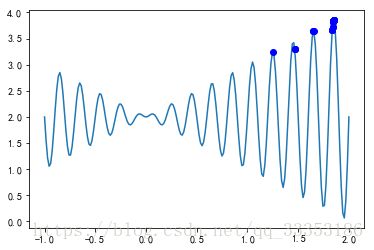
这是在Matlab的gatbx工具箱下实现简单遗传算法搜索f(x)=x*sin(10*pi*x)+2.0,在[-1,2]上的最大值的脚本程序,运行结果如下:
result =
3.8500
时间已过 0.081932 秒。
再看一下在Geatpy上如何编写脚本:
-
"""demo.py"""
-
import numpy
as np
-
import geatpy
as ga
# 导入geatpy库
-
import matplotlib.pyplot
as plt
-
import time
-
-
"""============================目标函数============================"""
-
def aim(x):
# 传入种群染色体矩阵解码后的基因表现型矩阵
-
return x * np.sin(
10 * np.pi * x) +
2.0
-
x = np.linspace(
-1,
2,
200)
-
plt.plot(x, aim(x))
# 绘制目标函数图像
-
start_time = time.time()
# 开始计时
-
"""============================变量设置============================"""
-
x1 = [
-1,
2]
# 自变量范围
-
b1 = [
1,
1]
# 自变量边界
-
codes = [
1]
# 变量的编码方式,2个变量均使用格雷编码
-
precisions =[
5]
# 变量的精度
-
scales = [
0]
# 采用算术刻度
-
ranges=np.vstack([x1]).T
# 生成自变量的范围矩阵
-
borders=np.vstack([b1]).T
# 生成自变量的边界矩阵
-
"""========================遗传算法参数设置========================="""
-
NIND =
40;
# 种群个体数目
-
MAXGEN =
25;
# 最大遗传代数
-
GGAP =
0.9;
# 代沟:说明子代与父代的重复率为0.1
-
"""=========================开始遗传算法进化========================"""
-
FieldD = ga.crtfld(ranges,borders,precisions,codes,scales)
# 调用函数创建区域描述器
-
Lind = np.sum(FieldD[
0, :])
# 计算编码后的染色体长度
-
Chrom = ga.crtbp(NIND, Lind)
# 根据区域描述器生成二进制种群
-
variable = ga.bs2rv(Chrom, FieldD)
#对初始种群进行解码
-
ObjV = aim(variable)
# 计算初始种群个体的目标函数值
-
pop_trace = (np.zeros((MAXGEN,
2)) * np.nan)
# 定义进化记录器,初始值为nan
-
ind_trace = (np.zeros((MAXGEN, Lind)) * np.nan)
# 定义种群最优个体记录器,记录每一代最优个体的染色体,初始值为nan
-
# 开始进化!!
-
for gen
in range(MAXGEN):
-
FitnV = ga.ranking(-ObjV)
# 根据目标函数大小分配适应度值(由于遵循目标最小化约定,因此最大化问题要对目标函数值乘上-1)
-
SelCh=ga.selecting(
'sus', Chrom, FitnV, GGAP)
# 选择
-
SelCh=ga.recombin(
'xovsp', SelCh,
0.7)
# 重组(采用单点交叉方式,交叉概率为0.7)
-
SelCh=ga.mutbin(SelCh)
# 二进制种群变异
-
variable = ga.bs2rv(SelCh, FieldD)
# 对育种种群进行解码(二进制转十进制)
-
ObjVSel = aim(variable)
# 求育种个体的目标函数值
-
[Chrom,ObjV] = ga.reins(Chrom,SelCh,SUBPOP,
2,
1,ObjV,ObjVSel)
# 重插入得到新一代种群
-
# 记录
-
best_ind = np.argmax(ObjV)
# 计算当代最优个体的序号
-
pop_trace[gen,
0] = ObjV[best_ind]
# 记录当代种群最优个体目标函数值
-
pop_trace[gen,
1] = np.sum(ObjV) / ObjV.shape[
0]
# 记录当代种群的目标函数均值
-
ind_trace[gen, :] = Chrom[best_ind, :]
# 记录当代种群最优个体的变量值
-
# 进化完成
-
end_time = time.time()
# 结束计时
-
"""============================输出结果及绘图================================"""
-
print(
'目标函数最大值:',np.max(pop_trace[:,
0]))
# 输出目标函数最大值
-
variable = ga.bs2rv(ind_trace, FieldD)
# 解码得到表现型
-
print(
'用时:', end_time - start_time)
-
plt.plot(variable, aim(variable),
'bo')
运行结果如下:
目标函数最大值: 3.850273756279405
用时: 0.0509946346282959
对比发现,Geatpy的运行效率要高于Matlab,而且结果较好。
对比Matlab代码和Python代码,我们会发现Geatpy提供风格极为相似的库函数,有Matlab相关编程经验的基本上可以无缝转移到Python上利用Geatpy进行遗传算法程序开发。
2. 利用框架实现遗传算法。
(注意:Geatpy已于2018.08.27更新至1.0.4版本,更新了内置的多目标优化算法编程模板。下面的代码最后一行调用编程模板的返回参数不再是3个而是4个,并且传入的参数有所变化。请执行pip install --upgrade geatpy 将电脑中的geatpy更新至最新版本。)
Geatpy提供开放的进化算法框架。即“函数接口”+“编程模板”。对于一些复杂的进化算法,如多目标进化优化、改进的遗传算法等,利用上面所说的编写脚本是非常麻烦的,改用框架的方法可以极大提高编程效率。
这里给出一个利用框架实现NSGA-II算法求多目标优化函数ZDT-1的帕累托前沿面的例子:
首先编写函数接口文件:
-
""" aimfuc.py """
-
-
# ZDT1
-
def aimfuc(Chrom):
-
ObjV1 = Chrom[:,
0]
-
gx =
1 + (
9 /
29) * np.sum(Chrom[:,
1:
30],
1)
-
hx =
1 - np.sqrt(ObjV1 / gx)
-
ObjV2 = gx * hx
-
return np.array([ObjV1, ObjV2]).T
然后编写脚本,使用Geatpy提供的nsga2算法的编程模板(nsga2_templet):
-
"""main.py"""
-
import numpy
as np
-
import geatpy
as ga
# 导入geatpy库
-
-
# 获取函数接口地址
-
AIM_M = __import__(
'aimfuc')
-
AIM_F =
'aimfuc'
-
-
"""============================变量设置============================"""
-
ranges = np.vstack([np.zeros((
1,
30)), np.ones((
1,
30))])
# 生成自变量的范围矩阵
-
borders = np.vstack([np.ones((
1,
30)), np.ones((
1,
30))])
# 生成自变量的边界矩阵
-
precisions = [
4] *
30
# 自变量的编码精度
-
"""========================遗传算法参数设置========================="""
-
NIND =
25
# 种群规模
-
MAXGEN =
1000
# 最大遗传代数
-
GGAP =
1;
# 代沟:子代与父代的重复率为(1-GGAP),由于后面使用NSGA2算法,因此该参数无用
-
selectStyle =
'tour'
# 遗传算法的选择方式
-
recombinStyle =
'xovdprs'
# 遗传算法的重组方式,设为两点交叉
-
recopt =
0.9
# 交叉概率
-
pm =
0.1
# 变异概率
-
SUBPOP =
1
# 设置种群数为1f
-
maxormin =
1
# 设置标记表明这是最小化目标
-
MAXSIZE =
1000
# 帕累托最优集最大个数
-
FieldDR = ga.crtfld(ranges, borders, precisions)
# 生成区域描述器
-
"""=======================调用编程模板进行种群进化==================="""
-
# 得到帕累托最优解集NDSet以及解集对应的目标函数值NDSetObjV
-
[ObjV, NDSet, NDSetObjV, times] = ga.q_sorted_templet(AIM_M, AIM_F,
None,
None, FieldDR,
'R', maxormin, MAXGEN, MAXSIZE, NIND, SUBPOP, GGAP, selectStyle, recombinStyle, recopt, pm, drawing =
1)
运行结果如下:
用时: 7.359716176986694 秒
帕累托前沿点个数: 479 个
单位时间找到帕累托前沿点个数: 65 个
可以改用q_sorted_templet快速非支配排序的多目标优化编程模板,可以得到更好的效率和更好的结果:
进化算法的核心算法逻辑是写在编程模板内部的,代码是开源的,我们可以参考Geatpy编程模板的源代码来自定义自己的编程模板,以实现丰富多样的进化算法,如差分进化算法、改进的遗传算法等:
https://github.com/geatpy-dev/geatpy/tree/master/geatpy/source-code/templets
后面的博客将深入理解Geatpy的库函数用法,以及探讨框架的核心——编程模板的实现。还会讲一些使用Geatpy解决问题的案例。欢迎继续跟进~感谢!
下一篇:Python遗传和进化算法框架(二)Geatpy库函数和数据结构
https://blog.csdn.net/qq_33353186/article/details/82020507