QIIME 2教程. 23图形界面q2studio(2020.2)
文章目录
- 前情提要
- 图形界面`q2studio`
- Reference
- 译者简介
- 猜你喜欢
- 写在后面
前情提要
- NBT:QIIME 2可重复、交互式的微生物组分析平台
- 1简介和安装Introduction&Install
- 2插件工作流程概述Workflow
- 3老司机上路指南Experienced
- 4人体各部位微生物组分析Moving Pictures
- Genome Biology:人体各部位微生物组时间序列分析
- 5粪菌移植分析练习FMT
- Microbiome:粪菌移植改善自闭症
- 6沙漠土壤分析Atacama soil
- mSystems:干旱对土壤微生物组的影响
- 7帕金森小鼠教程Parkinson’s Mouse
- Cell:肠道菌群促进帕金森发生ParkinsonDisease
- 8差异丰度分析gneiss
- 9数据导入Importing data
- 10数据导出Exporting data
- 11元数据Metadata
- 12数据筛选Filtering data
- 13训练特征分类器Training feature classifiers
- 14数据评估和质控Evaluating and controlling
- 15样品分类和回归q2-sample-classifier
- 16纵向和成对样本比较q2-longitudinal
- 17鉴定和过滤嵌合体序列q2-vsearch
- 18序列双端合并read-joining
- 19使用q2-vsearch聚类OTUs
- 20实用程序Utilities
- 21进化树推断q2-phylogeny
- 22命令行界面q2cli
图形界面q2studio
QIIME 2 Studio (q2studio)
https://docs.qiime2.org/2020.2/interfaces/q2studio/
注:最好按本教程顺序学习,想直接学习本章,至少完成本系列《1简介和安装》。在此使用该教程中生成的
table.qza文件。
详者注:图形界面安装依赖关系较多,而且需要在图形界面环境下运行,一般服务器可能不支持。建议在VirtualBox预安装好的虚拟机Ubuntu的桌面环境下使用。
QIIME 2 Studio(q2studio)是QIIME 2的图形用户界面。本文档将向您展示如何安装QIIME 2 Studio并将其用作桌面应用程序。
注:QIIME 2 Studio当前需要源安装。 将来我们将提供软件包分发/安装程序,它将大大简化此处介绍的安装过程。 QIIME 2 Studio的实验版本可作为VirtualBox映像中的预安装包提供。
启动QIIME 2 Studio需要一个活动的conda环境,其中已安装QIIME 2。 无法激活兼容的conda环境可能会产生错误,并可能导致应用程序崩溃。 如果尚未激活conda环境,请立即进行以下操作:
source activate qiime2-2020.2
请注意,如果您或系统管理员在安装QIIME 2时提供了一个名称,则您的conda环境的名称可能不是qiime2-2020.2。
此接口要求您的系统具有Node.js。 我们目前需要版本5或更高版本; 您可以在此处找到安装说明。 将来不再需要这种依赖关系。
安装Node.js> = 5后,您需要下载并提取界面的源代码:
wget -c \
-O "q2studio-2020.2.0.zip" \
"https://codeload.github.com/qiime2/q2studio/zip/2020.2.0"
unzip q2studio-2020.2.0.zip && rm q2studio-2020.2.0.zip
cd q2studio-2020.2.0
接下来,我们需要安装它(既作为Python软件包,又作为Node.js软件包):
pip install .
npm install && npm run build
最后,我们将能够使用以下命令启动界面:
npm start
这将打开一个新窗口,如下所示:
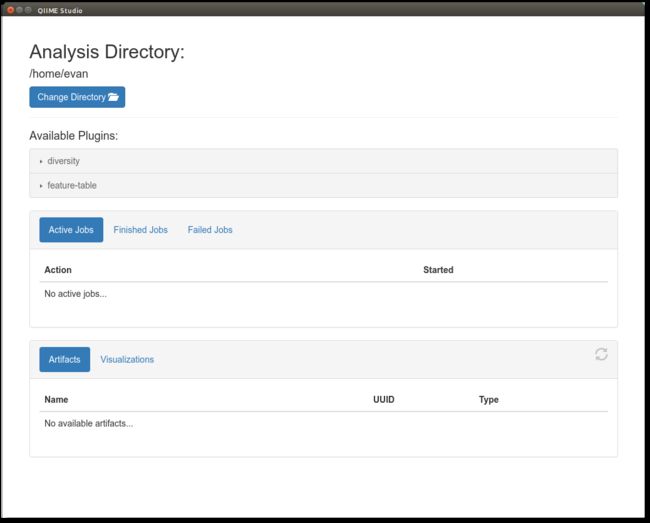
该界面分为四个部分。在顶部,您可以选择要在其中工作的目录。默认情况下,它从您的主目录开始,但是我们想将其更改为运行“移动图片 Moving Pictures tutorial ”教程的目录。为此,请单击“更改目录Change Directory”按钮并导航到该目录。
该界面的下一个组件是可用的插件。单击插件会显示可用的方法和可视化工具。请注意,这些是可通过命令行界面(和其他QIIME 2界面)使用的相同插件和方法/可视化工具。
在界面的中间,我们有作业窗格。您可以在活动作业Active Jobs,完成的作业Finished Jobs和失败的作业Failed Jobs之间选择。由于我们还没有运行任何内容,因此每个选项卡当前为空。
最后,在底部窗格中,您将在当前目录中看到工件和可视化效果。这些.qza和.qzv文件是从“移动图片 Moving Pictures tutorial ”教程生成的文件。
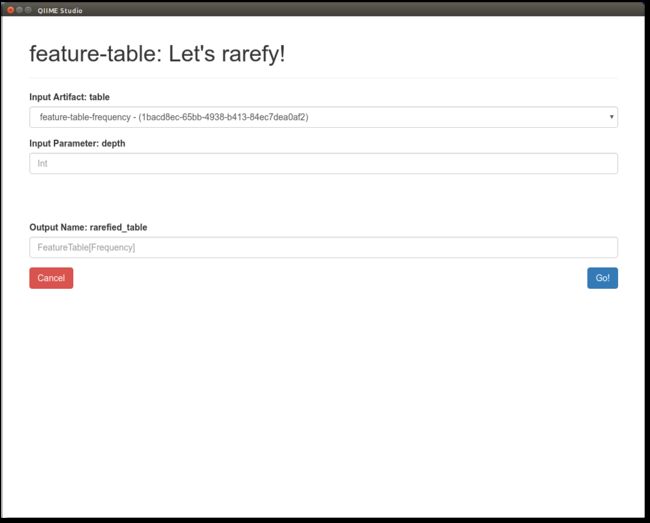
从feature-table插件中选择Rarefy table方法,这将更改页面,如下所示:
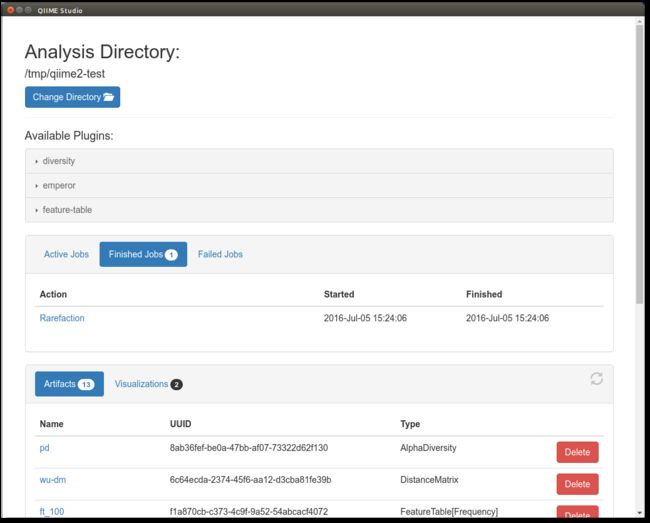
在此页面上,您将选择输入并命名输出。 与QIIME 2对象相对应的输入(Input Artifact: table:此方法)将填充有正确语义类型的可用对象。 选择在“人体各部位微生物组”教程中生成的table.qza对象作为输入。 填写稀疏深度(采样深度sampling depth)100,将输出文件命名为rarefied_table_100,然后单击GO执行! 这将启动一个作业,该作业将显示在“活动作业Active Jobs”选项卡中,直到完成为止,之后它将显示在“完成的作业Finished Jobs”选项卡中(如果出现问题,则显示在“失败的作业Failed Jobs”选项卡中)。
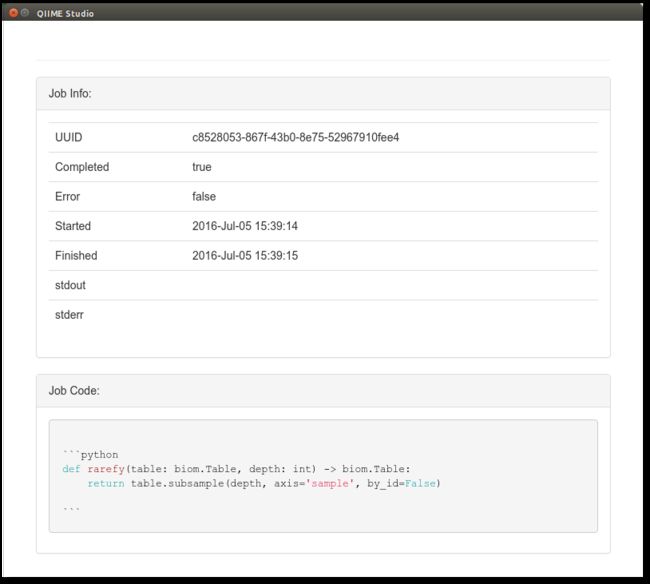
您可以通过单击来选择工作,这会打开一个新窗口,描述更多详细信息:
尝试使用QIIME 2 Studio尝试其他插件!
Reference
https://docs.qiime2.org/2020.2/
Evan Bolyen*, Jai Ram Rideout*, Matthew R. Dillon*, Nicholas A. Bokulich*, Christian C. Abnet, Gabriel A. Al-Ghalith, Harriet Alexander, Eric J. Alm, Manimozhiyan Arumugam, Francesco Asnicar, Yang Bai, Jordan E. Bisanz, Kyle Bittinger, Asker Brejnrod, Colin J. Brislawn, C. Titus Brown, Benjamin J. Callahan, Andrés Mauricio Caraballo-Rodríguez, John Chase, Emily K. Cope, Ricardo Da Silva, Christian Diener, Pieter C. Dorrestein, Gavin M. Douglas, Daniel M. Durall, Claire Duvallet, Christian F. Edwardson, Madeleine Ernst, Mehrbod Estaki, Jennifer Fouquier, Julia M. Gauglitz, Sean M. Gibbons, Deanna L. Gibson, Antonio Gonzalez, Kestrel Gorlick, Jiarong Guo, Benjamin Hillmann, Susan Holmes, Hannes Holste, Curtis Huttenhower, Gavin A. Huttley, Stefan Janssen, Alan K. Jarmusch, Lingjing Jiang, Benjamin D. Kaehler, Kyo Bin Kang, Christopher R. Keefe, Paul Keim, Scott T. Kelley, Dan Knights, Irina Koester, Tomasz Kosciolek, Jorden Kreps, Morgan G. I. Langille, Joslynn Lee, Ruth Ley, Yong-Xin Liu, Erikka Loftfield, Catherine Lozupone, Massoud Maher, Clarisse Marotz, Bryan D. Martin, Daniel McDonald, Lauren J. McIver, Alexey V. Melnik, Jessica L. Metcalf, Sydney C. Morgan, Jamie T. Morton, Ahmad Turan Naimey, Jose A. Navas-Molina, Louis Felix Nothias, Stephanie B. Orchanian, Talima Pearson, Samuel L. Peoples, Daniel Petras, Mary Lai Preuss, Elmar Pruesse, Lasse Buur Rasmussen, Adam Rivers, Michael S. Robeson, Patrick Rosenthal, Nicola Segata, Michael Shaffer, Arron Shiffer, Rashmi Sinha, Se Jin Song, John R. Spear, Austin D. Swafford, Luke R. Thompson, Pedro J. Torres, Pauline Trinh, Anupriya Tripathi, Peter J. Turnbaugh, Sabah Ul-Hasan, Justin J. J. van der Hooft, Fernando Vargas, Yoshiki Vázquez-Baeza, Emily Vogtmann, Max von Hippel, William Walters, Yunhu Wan, Mingxun Wang, Jonathan Warren, Kyle C. Weber, Charles H. D. Williamson, Amy D. Willis, Zhenjiang Zech Xu, Jesse R. Zaneveld, Yilong Zhang, Qiyun Zhu, Rob Knight & J. Gregory Caporaso#. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2. Nature Biotechnology. 2019, 37: 852-857. doi:10.1038/s41587-019-0209-9
译者简介
刘永鑫,博士。2008年毕业于东北农业大学微生物学专业,2014年于中国科学院大学获生物信息学博士学位,2016年中科院遗传发育所博士后出站留所任工程师。目前主要研究方向有微生物组数据分析、方法开发和科学传播。目前以第一作者(含共同)或微生物组数据分析负责人在Science、Nature Biotechnology、Cell Host & Microbe 等杂志发表论文20余篇,引用千余次。作为中国唯一单位代表参与微生物组分析平台QIIME 2开发。受邀以第一作者和/或通讯作者(含共同)在Protein & Cell、Current Opinion in Microbiology、遗传 等杂志发表微生物组研究方法综述。2017年7月创办“宏基因组”公众号,目前分享本领域相关原创文章1800余篇,代表作品有《微生物组图表解读、分析流程和统计绘图》、《QIIME2中文教程》等系列,关注人数9万+,累计阅读1400万+。
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA